·论 著·
武震东1,2,任洪波2,孙志强2,刘 斌2,牛国栋2,焦保华1*
(1.河北医科大学第二医院神经外科,河北 石家庄 050000;2.河北省邯郸市中心医院神经外科,河北 邯郸 056000)
[摘要] 目的探讨miR-34c对人脑胶质瘤细胞株U251和U87增殖、凋亡和细胞周期的影响。方法应用脂质体将miR-34c导入胶质瘤细胞株U251和U87后,应用定量PCR验证转染效果,应用CCK-8法检测细胞增殖变化,流式细胞仪检测细胞凋亡和细胞周期的改变。结果在U87和U251细胞系中,miR-34c-3p和miR-34c-5p的表达量明显小于正常胶质细胞系HEB。在U251细胞中,转染miR-34c-3p或miR-34c-5p后48、72、96 h,细胞存活率显著低于空白组和阴性对照组(P<0.05)。而在U87细胞中情况有所不同,转染miR-34c-3p后48、72、96 h,细胞存活率显著低于空白组和阴性对照组(P<0.05);转染miR-34c-5p后24、48、72、96 h,细胞存活率显著低于空白组(P<0.05),而与阴性对照组差异无统计学意义(P>0.05)。U251细胞高表达miR-34c-3p或miR-34c-5p,U87细胞高表达miR-34c-3p可以导致细胞停滞在S期,同时降低了G0/G1期的细胞数。在U251细胞中,凋亡细胞在空白组和阴性对照组分别占6.57%和6.3%,而转染miR-34c-3p或miR-34c-5p后凋亡细胞所占比例上升分别占28.49%和28.14%。但是在U87细胞中有所不同,仅转染miR-34c-3p后凋亡细胞数上升(3.56%),而空白组、阴性对照组和转染miR-34c-5p组分别为1.53%、1.68%和1.91%。结论转染上调miR-34c在胶质瘤中的表达能够抑制其增殖,导致细胞周期在S期及G2/M期的停滞,诱导胶质瘤细胞的凋亡,为脑胶质瘤靶向治疗提供可靠的依据。但miR-34c-3p与miR34c-5p在不同细胞系中,影响细胞增殖能力、细胞周期变化及诱导细胞凋亡的效果有所不同,推测它们可能是通过不同靶点或机制起作用。
[关键词] 神经胶质瘤;微RNAs;细胞增殖;细胞凋亡 doi:10.3969/j.issn.1007-3205.2015.08.024
[收稿日期]2015-06-11;[修回日期]2015-07-21
[基金项目]河北省医学科学研究重点课题(ZD20140280)
[作者简介]武震东(1974-),男,辽宁葫芦岛人,河北省邯郸市中心医院副主任医师,医学博士,从事神经外科疾病诊治研究。
通讯作者*。E-mail:jiaobp000@163.com
[中图分类号] R730.264 [文献标志码] B
微小RNA(microRNA,miRNA)是一种约22个核苷酸长度非编码单链小RNA,其本身不具有翻译蛋白质的功能,通过与mRNA 的3′端非翻译区(3′UTR)碱基序列互补配对在转录后水平调控靶基因表达。miRNA与mRNA 能进行完全或近乎完全的互补配对,诱导对mRNA 的切割降解,而部分碱基配对则导致功能蛋白质的转录抑制[1]。最近有报道miRNA与肿瘤恶性程度及肿瘤患者的预后有密切关系[2]。亦有报道与正常组织比较,恶性肿瘤组织的miRNA异常表达[3]。MicroRNA-34c(miR-34c)在胶质瘤组织中广泛低表达,且随胶质瘤级别的升高,miR-34c-3p和miR-34c-5p的表达显著降低,miR-34c-3p和miR-34c-5p表达与胶质瘤级别呈负相关。本研究为胶质瘤细胞系U251和U87细胞转染miR-34c-3p和miR-34c-5p,上调其表达,检测miR-34c对人脑胶质瘤细胞增殖、细胞周期、凋亡等生物学特性的影响,旨在为胶质瘤的治疗开拓新的思路。
1.1 细胞株 胶质瘤细胞系U251和U87细胞购于中国科学院细胞库。正常人胶质细胞系HEB细胞购自中国科学院广州生物医药与健康研究院。
1.2 实验方法
1.2.1 细胞培养 U251用含10%胎牛血清(fetal calf serum,FBS)的DMEM-F12培养基,U87和HEB用含10%FBS的DMEM-高糖培养基培养,将符合细胞计数要求的细胞悬液分装入培养瓶内,将培养瓶放入37 ℃和5%CO2的培养箱内24~48 h后换液继续培养,换液的时间由细胞贴壁情况而定。培养U251、U87、HEB细胞达到指数生长期进行实验。利用脂质体介导的转染方法,将化学合成的miR-34c寡核苷酸转染到胶质瘤U87和U251细胞中,同时设立转染非特异性寡核苷酸片段的阴性对照组,使miR-34c基因有效的过表达。将未转染任何核苷酸链的胶质瘤U87和U251细胞为空白组。
1.2.2 细胞转染miRNA 转染采用脂质体转染试剂LipofectamineTM RNAiMAX转染试剂,用LipofectamineTM RNAiMAX转染miRNA(化学合成的成熟miRNA模拟物mimics浓度为50 nmol/L)到细胞内,转染时使用Opti-MEM培养基,转染后4 h换为细胞生长培养基(U251用含10%FBS的DMEM-F12培养基,U87和HEB用含10%FBS的DMEM-高糖培养基)。
1.2.3 定量PCR检测转染效果 利用RNA抽提试剂Trizol(Invitrogen, USA)提取收集细胞总RNA。取1 μg总RNA做茎环引物的逆转录反应。miR-34c-3p和 miR-34c-5p的共同逆转录引物为miRNA R:5′ CTCAACTGGTGTCGTGGA。PCR引物为hsa-miR-34c-3p F:5′ ACACTCCAGCTG-GGAATCACTAACCACACGG;hsa-miR-34c-5p F:5′ACACTCCAGCTGGGAGGCAGTGTAGTT-AGCTGAT。内参U6的引物为U6-F:5′CTCGC-TTCGGCAGCACA;U6-R:5′AACGCTTCACG-AATTTGCGT。所有引物均为上海生工生物工程公司合成。使用的定量PCR仪为ABI PRISM® 7500 Sequence Detection System。PCR扩增程序如下:95 ℃变形5 min,随后95 ℃作用15 s,65 ℃作用15 s,72 ℃作用32 s,共进行40个循环,在温度60~95 ℃进行融解曲线分析,每个样本重复3次。
1.2.4 细胞增殖-毒性检测(Cell Counting Kit-8,CCK-8)法检测细胞增殖能力 取转染后细胞进行下面实验。吹打散细胞,调整细胞浓度为1×105个/mL,分到96孔板,每孔100 μL,即每孔细胞为1×104个。等细胞半贴壁完全,收集各个时间点的细胞24、48、72、96 h加入CCK-8溶液细胞增殖检测试剂,比例为1∶10。即100 μL培养液加入10 μL检测液。在孵育4 h后,酶标仪读板光密度(optical delnsity,OD)490数据。细胞存活率=OD转染组/OD对照组。
1.2.5 细胞周期检测 细胞转染48 h后每样收集1×106细胞。离心收集细胞,弃上清,用预冷PBS洗细胞2次,加入预冷70%乙醇,于4 ℃固定过夜,或-20 ℃长期固定。离心收集细胞,以1 mL的PBS洗细胞1次,加入500 μL PBS含50 μg/mL溴化丙锭(propidiumiodide,PI),100 μg/mL核糖核酸酶A(Ribonuclease A,RNase A),0.2%聚乙二醇辛基苯基醚(Triton X-100),4 ℃避光孵育30 min。以标准程序用流式细胞仪检测,一般计数2~3万个细胞,结果用细胞周期拟和软件ModFit分析。
1.2.6 细胞凋亡检测 细胞转染48 h后,将细胞培养板中各组细胞的培养基分别转移到15 mL的锥形管中并置于冰上。用2 mL PBS溶液轻轻润洗培养板内细胞,去除PBS溶液,加入0.5 mL 0.25%不含EDTA的胰酶孵育,直到显微镜下观察到细胞开始从培养板壁脱落。轻轻连续拍打使细胞从培养板壁上完全脱落,将细胞轻轻重悬于预冷的1×结合缓冲液中使得其密度为1×106细胞/mL,将0.5 mL细胞悬液从细胞培养板中(5×105个细胞)转移到1个干净的离心管内,加入1.25 μL 细胞凋亡检测试剂Annexin V-FITC,室温(18~24 ℃)避光反应15 min,室温1 000 g 离心5 min,去除上清,将细胞用0.5 mL预冷的1×结合缓冲液轻轻重悬,加入10 μL PI,立即用流式细胞仪检测分析。
1.3 统计学方法 应用SPSS 16.0统计学软件进行数据分析。计量资料以![]() ±s表示,分别采用单因素方差分析、LSD-t检验和重复测量设计资料的方差分析。P<0.05为差异有统计学意义。
±s表示,分别采用单因素方差分析、LSD-t检验和重复测量设计资料的方差分析。P<0.05为差异有统计学意义。
2.1 定量PCR检测miR-34c-3p和miR-34c-5p的转染效果 在U87和U251细胞系中,miR-34c-3p和miR-34c-5p的表达量要明显小于正常胶质细胞系HEB(图1,2)。而转染了miR-34c-3p或miR-34c-5p的U87和U251细胞,其miR-34c-3p的表达量明显高于空白组(P<0.05,图1)和阴性对照(P<0.05)。该结果确定了miRNA确实转染成功。
2.2 转染miR-34c抑制胶质瘤细胞的增殖能力 在U251细胞中,转染miR-34c-3p或miR-34c-5p后48、72、96 h,细胞存活率显著低于空白组(P<0.05)和阴性对照组(P<0.05)(图3)。而在U87细胞中情况有所不同,转染miR-34c-3p后48、72、96 h,细胞存活率显著低于空白组(P<0.05)和阴性对照组(P<0.05);转染miR-34c-5p后24、48、72、96 h,细胞存活率显著低于空白组(P<0.05),而与阴性对照组差异无统计学意义(P>0.05)(图4)。
2.3 转染miRNA-34c后细胞周期的变化 在U251细胞中,空白组和阴性对照组细胞的G0/G1期分别占72.7%和73.44%,而转染miR-34c-3p或miR-34c-5p后,其G0/G1期分别降至44.24%和43.58%。而空白组和阴性对照组细胞的G2/M期分别仅占4.4%和4.31%,转染miR-34c-3p或miR-34c-5p后,其G2/M期分别升至21.6%和21.83%。S期与G2/M期相似,转染后所占比例上升(图5)。在U87细胞中,空白组和阴性对照组细胞的G0/G1期分别占69.3%和75.62%,转染miR-34c-3p后,其G0/G1期降至44.05%,而转染miR-34c-5p却不能影响G0/G1期(79.07%)。空白组和阴性对照组细胞的S期分别占20.66%和13.61%,转染miR-34c-3p后,其S期升至48.83%,但转染miR-34c-5p同样不能影响S期(11.04%)。G2/M期在4组中所占比例差异无统计学意义(图5)。该实验证实了U251细胞高表达miR-34c-3p或miR-34c-5p,U87细胞高表达miR-34c-3p可以导致细胞停滞在S期,同时降低了G0/G1期的细胞数。细胞周期的变化说明miR-34c可能通过抑制细胞周期中的某个通路以达到抑制细胞增殖的目的。
2.4 转染miRNA-34c后诱导胶质瘤细胞的凋亡 根据流式细胞仪的结果,Annexin V和PI双阳性的细胞即为凋亡细胞。在U251细胞中,凋亡细胞在空白组和阴性对照组分别占6.57%和6.3%,而转染miR-34c-3p或miR-34c-5p后凋亡细胞所占比例上升分别占28.49%和28.14%。但是在U87细胞中有所不同,仅转染miR-34c-3p后凋亡细胞数上升(3.56%),而空白组,阴性对照组和转染miR-34c-5p组分别为1.53%,1.68%和1.91%(图6)。
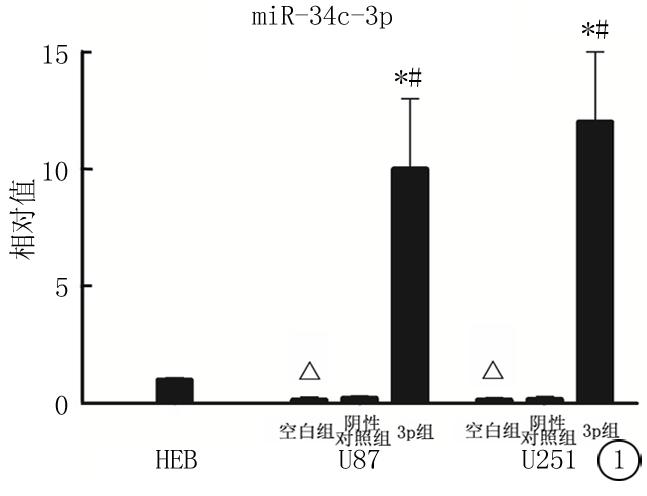
图1 实时定量PCR检测正常胶质细胞系HEB、U251和U87细胞系中miR-34c-3p的表达
*P<0.05为miR-34c-3p组与空白组比较 #P<0.05为miR-34c-3p组与阴性对照组比较 △P<0.05为空白组与HEB比较(LSD-t检验)
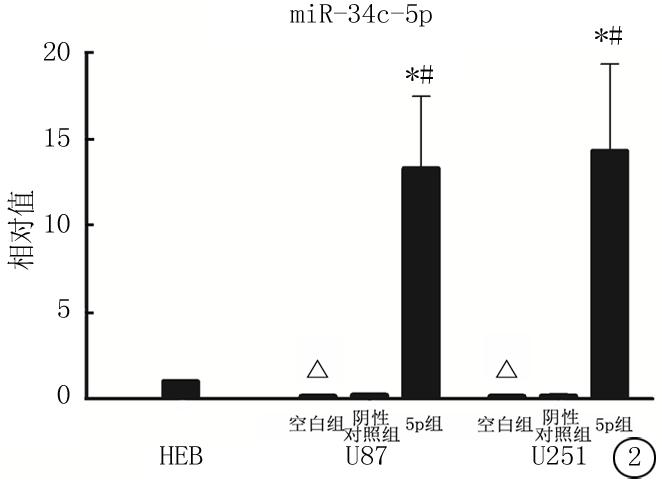
图2 实时定量PCR检测正常胶质细胞系HEB、U251和U87细胞系中miR-34c-5p的表达
*P<0.05为miR-34c-5p组与空白组比较 #P<0.05为miR-34c-5p组与阴性对照组比较 △P<0.05为空白组与HEB比较(LSD-t检验)
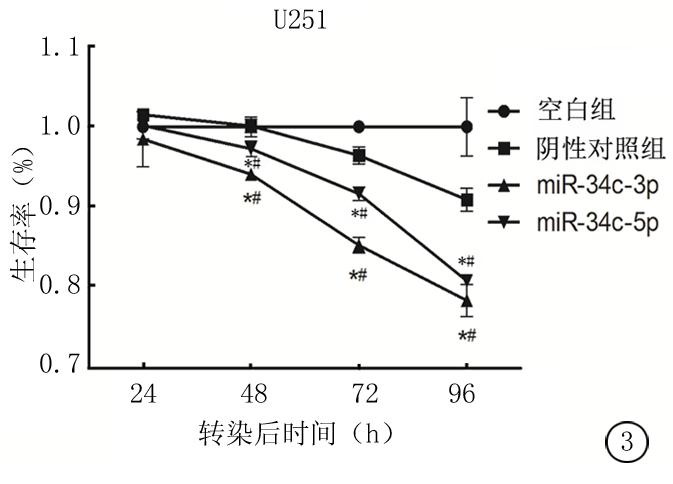
图3 转染miR-34c-3p或miR-34c-5p后,U251细胞的生存率
*P<0.05为转染组与空白组比较 #P<0.05为转染组与阴性对照组比较(重复测量设计资料的方差分析)
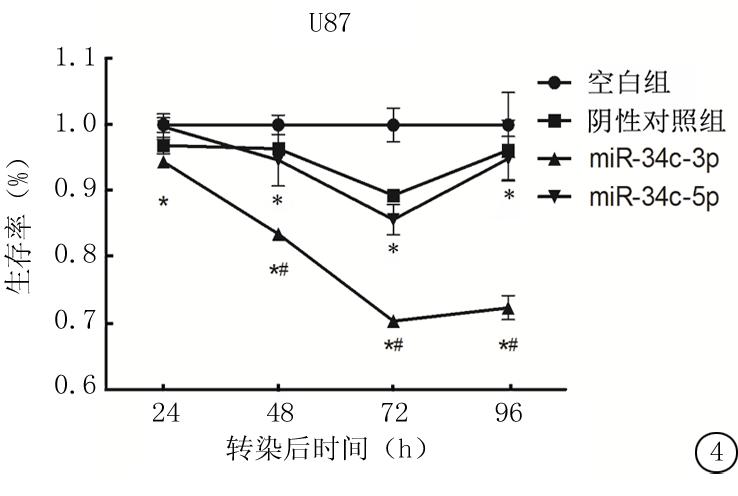
图4 转染miR-34c-3p或miR-34c-5p后,U87细胞的生存率
*P<0.05为转染组与空白组比较 #P<0.05为转染组与阴性对照组比较(重复测量设计资料的方差分析)
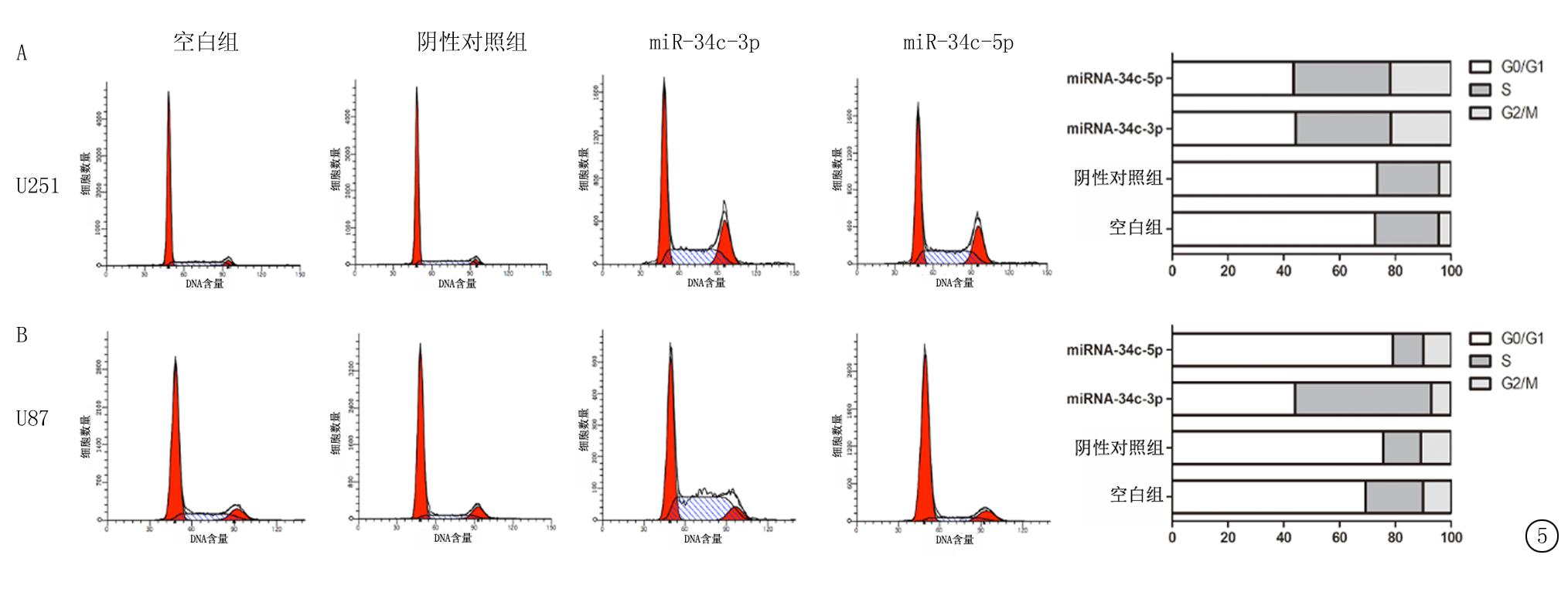
图5 转染miRNA-34c-3p或miR-34c-5p后,细胞的细胞周期情况
A.U251细胞;B.U87细胞
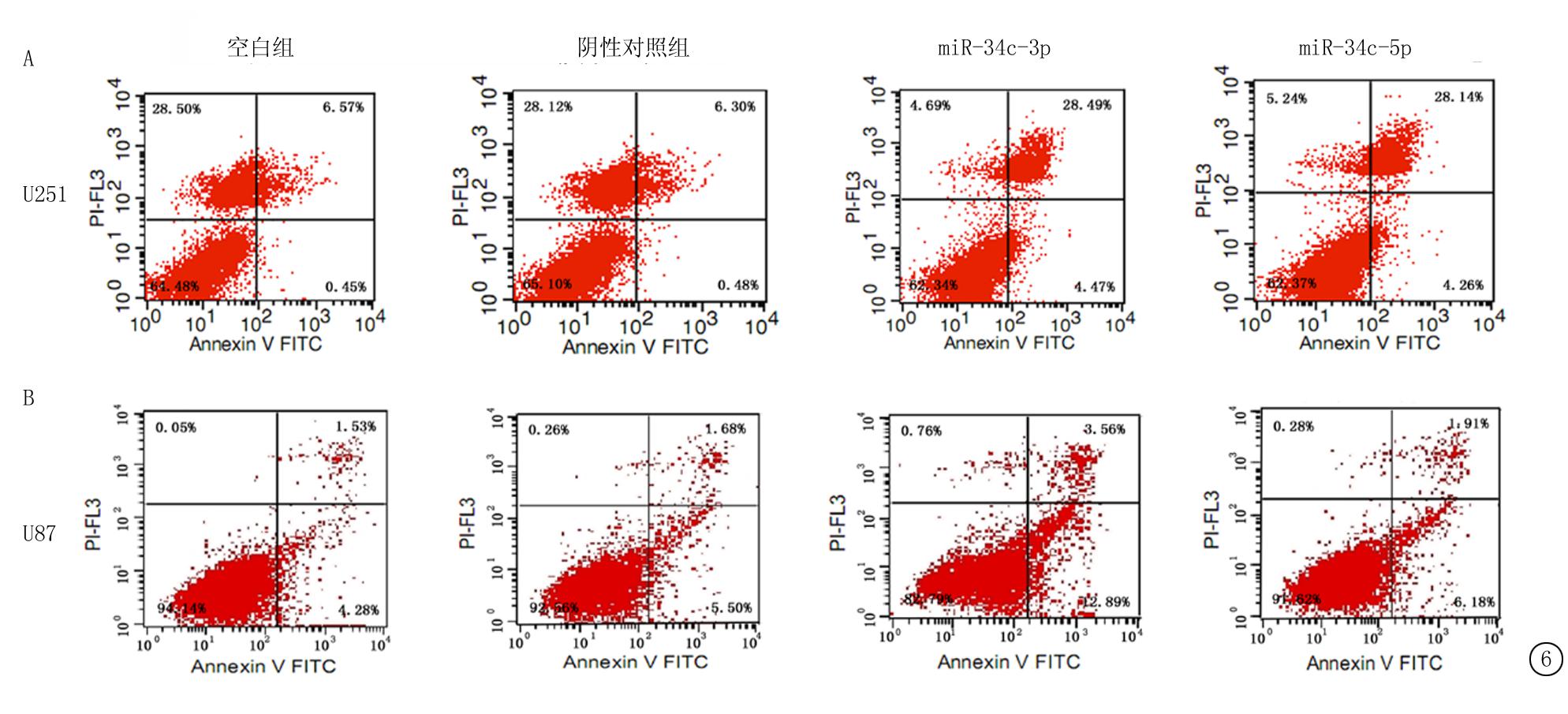
图6 转染miR-34c-3p或miR-34c-5p后,细胞的凋亡情况
A.U251细胞;B.U87细胞
肿瘤的发生是细胞增殖、凋亡和分化失衡的结果。越来越多的研究证实许多miRNA具有促进细胞增殖和存活的作用,同时还有许多miRNA具有抑制细胞增殖和存活的作用,这两类miRNA作为癌基因和抑癌基因在肿瘤的发生发展过程中发挥着重要的作用[4-5]。说明将miRNA作为肿瘤治疗的靶标是可行的。在近期的研究中,miR-34异常表达抑制细胞分化、克隆形成,使细胞周期阻滞在G1期[6-7]。并且,miR-34a的再表达诱导了细胞的凋亡[8]。miR-34介导的细胞凋亡能够被p53基因的失活所抑制。miR-34a靶向一些mRNA,如SIRT1(Sirtuin type 1)、Bcl-2(B-cell lymphoma-2)、N-myc、细胞周期蛋白D1(cyclin D1),抑制这些基因转录[6,9-10]。最近有报道,miR-34c能够抑制血管平滑肌细胞的增殖[11]。
恶性胶质瘤是神经系统常见的肿瘤。miR-34c在人类恶性胶质瘤组织和多种细胞系中低表达。本研究利用多种细胞生物学方法,进一步探讨miR-34c对胶质瘤U87和U251细胞系的生物学功能,以便为胶质瘤的治疗寻找新的靶标。本研究首先检测转基因细胞对胶质瘤细胞增殖的影响,利用经典的CCK-8实验对细胞进行检测,结果显示转染了miR-34c-3p或miR-34c-5p的细胞能够明显减弱细胞的增殖,但U87细胞中miR-34c-5p与其阴性对照组比较差异无统计学意义,也就是说miR-34c-5p的抑制增殖作用比较弱,猜测在U87细胞中miR-34c-5p对细胞的抑制机制可能与miR-34c-3p有所不同。细胞周期是指一个母细胞分裂形成的细胞到下一次再分裂形成2个子细胞的时期,分为间期(G1、S、G2期)和有丝分裂期(M期)。本研究采用流式细胞仪分析转染miR-34c-3p或miR-34c-5p后细胞周期的变化,发现转染后的U251细胞多停滞在S期及G2/M期,而仅转染了miR-34c-3p的U87细胞多停滞在S期,转染了miR-34c-5p的U87细胞周期变化与空白组和阴性对照组差异无统计学意义,表明miR-34c-5p抑制U87细胞的机制与miR-34c-3p不同,这也进一步佐证了细胞增殖能力的结果。凋亡细胞对所有用于细胞活性鉴定的染料,如PI有抗染性,细胞膜有损伤的细胞的DNA可被PI着染产生红色荧光,而细胞膜保持完好的细胞则不会有红色荧光产生。本研究结果显示,转染miR-34c-3p或miR-34c-5p的U251细胞的凋亡率明显增高,而转染miR-34c-3p或miR-34c-5p的U87细胞凋亡率变化不大。
从体外实验数据看出,miR-34c-3p和miR-34c-5p能够抑制胶质瘤细胞增殖,导致细胞周期停滞在S期及G2/M期,诱导胶质瘤细胞的凋亡,大胆推测miR-34c-3p和miR-34c-5p在胶质瘤中扮演抑癌的角色,但miR-34c-3p与miR-34c-5p在不同细胞系中,影响细胞增殖能力、细胞周期变化及诱导细胞凋亡的效果有所不同,推测它们可能是通过不同靶点或机制起作用,有待于在以后的实验中继续探讨miR-34c-3p和miR-34c-5p在胶质瘤的作用机制。
[参考文献]
[1] Nelson KM,Weiss GJ.MicroRNAs and cancer:past,present,and potential future[J].Mol Cancer Ther,2008,7(12):3655-3660.
[2] Gao H,Zhao H,Xiang W.Expression level of human miR-34a correlates with glioma grade and prognosis[J].J Neurooncol,2013,113(2):221-228.
[3] Croce CM.Causes and consequences of microRNA dysregulation in cancer[J].Nat Rev Genet,2009,10(10):704-714.
[4] Deng S,Calin GA,Croce CM,et al.Mechanisms of microRNA deregulation in human cancer[J].Cell Cycle,2008,7(17):2643-2646.
[5] Medina PP,Slack FJ.microRNAs and cancer:an overview[J].Cell Cycle,2008,7(16):2485-2492.
[6] Bommer GT,Gerin I,Feng Y,et al.p53-mediated activation of miRNA34 candidate tumor-suppressor genes[J].Curr Biol,2007,17(15):1298-1307.
[7] Tarasov V,Jung P,Verdoodt B,et al.Differential regulation of microRNAs by p53 revealed by massively parallel sequencing:miR-34a is a p53 target that induces apoptosis and G1-arrest[J].Cell Cycle,2007,6(13):1586-1593.
[8] Hermeking H.The miR-34 family in cancer and apoptosis[J].Cell Death Differ,2010,17(2):193-199.
[9] Yamakuchi M,Ferlito M,Lowenstein CJ.miR-34a repression of SIRT1 regulates apoptosis[J].Proc Natl Acad Sci U S A,2008,105(36):13421-13426.
[10] Christoffersen NR,Shalgi R,Frankel LB,et al.p53-independent upregulation of miR-34a during oncogene-induced senescence represses MYC[J].Cell Death Differ,2010,17(2):236-245.
[11] Choe N,Kwon JS,Kim YS,et al.The microRNA miR-34c inhibits vascular smooth muscle cell proliferation and neointimal hyperplasia by targeting stem cell factor[J].Cell Signal,2015,27(6):1056-1065. KM,Weiss GJ.MicroRNAs and cancer:past,present,and potential future[J].Mol Cancer Ther,2008,7(12):3655-3660.
[2] Gao H,Zhao H,Xiang W.Expression level of human miR-34a correlates with glioma grade and prognosis[J].J Neurooncol,2013,113(2):221-228.
[3] Croce CM.Causes and consequences of microRNA dysregulation in cancer[J].Nat Rev Genet,2009,10(10):704-714.
[4] Deng S,Calin GA,Croce CM,et al.Mechanisms of microRNA deregulation in human cancer[J].Cell Cycle,2008,7(17):2643-2646.
[5] Medina PP,Slack FJ.microRNAs and cancer:an overview[J].Cell Cycle,2008,7(16):2485-2492.
[6] Bommer GT,Gerin I,Feng Y,et al.p53-mediated activation of miRNA34 candidate tumor-suppressor genes[J].Curr Biol,2007,17(15):1298-1307.
[7] Tarasov V,Jung P,Verdoodt B,et al.Differential regulation of microRNAs by p53 revealed by massively parallel sequencing:miR-34a is a p53 target that induces apoptosis and G1-arrest[J].Cell Cycle,2007,6(13):1586-1593.
[8] Hermeking H.The miR-34 family in cancer and apoptosis[J].Cell Death Differ,2010,17(2):193-199.
[9] Yamakuchi M,Ferlito M,Lowenstein CJ.miR-34a repression of SIRT1 regulates apoptosis[J].Proc Natl Acad Sci U S A,2008,105(36):13421-13426.
[10] Christoffersen NR,Shalgi R,Frankel LB,et al.p53-independent upregulation of miR-34a during oncogene-induced senescence represses MYC[J].Cell Death Differ,2010,17(2):236-245.
[11] Choe N,Kwon JS,Kim YS,et al.The microRNA miR-34c inhibits vascular smooth muscle cell proliferation and neointimal hyperplasia by targeting stem cell factor[J].Cell Signal,2015,27(6):1056-1065.
(本文编辑:赵丽洁)
![]()
参考文献重申对文后著录的要求
参考文献本刊作者在撰稿时请按照《<河北医科大学学报>投稿须知》中所列各项办理。特别是文后的著录,应以中华人民共和国国家标准(GB/T 7714-2005)为准。参考文献顺序号用阿拉伯数字加方括号标记;文献 的作者,1~3名需全部列出,3名以上只列前3名,后加“等”或“et al”、“他”;列出文献的题目(书籍列书名)和期刊名称(书籍列版次、出版地、出版社名);期刊列出年份、卷次和期次以及起-止页码(书籍列出年份和起-止页码)。缺少任何一项,均为不规范。
《河北医科大学学报》编辑部