
·综 述·
黄耀孟1,吴 珺1(综述),李秉慧2*(审校)
(1.河北医科大学生物化学与分子生物学教研室,河北 石家庄 050017;2. 河北医科大学第四医院外科, 河北 石家庄 050011)
[关键词]乳腺肿瘤;微RNAs;标志物 doi:10.3969/j.issn.1007-3205.2018.09.027
乳腺癌是世界上最常见的恶性疾病之一,每年估计有150万例新发病例[1],严重影响人类的健康。由于缺乏敏感性和特异性俱佳的生物标志物,故尚不能实现对乳腺癌的早期诊断和治疗。外周血循环核酸在乳腺癌诊断、预后和监测抗癌治疗反应中起重要作用。鉴于微小RNA(microRNA,miRNA)的易分离性和稳定性的特点,循环miRNA受到了越来越多的关注。现就miRNA在乳腺癌诊断、预后和预测中的应用综述如下。
1.1miRNA miRNA是含19~23个核苷酸的短单链RNA序列,miRNA在不同种类癌症组织中均有表达的异常变化。miRNA通过对靶基因3′非翻译区中的互补序列进行碱基配对介导靶基因抑制,导致转录物不稳定、翻译抑制等情况。 最近的研究报道,miRNA也可以通过与其他区域(包括蛋白质编码的外显子)结合调节基因表达[2],甚至可以诱导哺乳动物细胞中的基因表达。
1.2miRNA在乳腺癌中的作用 根据miRNA在乳腺癌中的表达变化及作用将其分为抑癌miRNA和原癌miRNA。let-7是已知的抑癌miRNA,在肿瘤组织中经常表达下调;其靶向定位LIN28,并且自身是LIN28负反馈调节的靶标。LIN28蛋白在包括乳腺癌在内的许多肿瘤中表达上调[3]。还有研究发现let-7通过靶向调节H-ras基因和高迁移率族蛋白(high mobility group protein,HMG)A调节乳腺癌肿瘤激活细胞(tumor-initiating cells,T-IC)2[4]。 miR-200家族也具有肿瘤抑制作用[5]。该家族由5个成员组成,分别组成1和12号染色体上的2个簇, 即簇Ⅰ(miR-200b/200a/429)和簇Ⅱ(miR-200c/141),其在上皮细胞间质转型(epithelial-mesenchymal transition,EMT)时被抑制,这与乳腺癌细胞迁移和侵袭性增加有关。miR-200家族成员在乳腺癌激活细胞中调节B细胞特异的莫罗尼白血病插入位点1基因的表达,并且通过抑制E盒结合锌指蛋白(zinc finger E-box binding hemeobox,ZEB)1和2抑制EMT。另有研究表明在乳腺癌细胞中,miR-200c的调控会影响细胞迁移和侵袭,miR-200c通过靶向定位肌动蛋白调节物,蛋白磷酸酶和Mg2+/Mn2+-依赖性因子(Mg2+/Mn2+-dependent factor,PPMF)1调节独立于ZEB/E-钙黏蛋白轴的转化生长因子β诱导的压力纤维蛋白的形成[6]。MiR-335通常在乳腺癌中沉默,通过靶向SRY相关的高迁移率族蛋白转录因子4和细胞外基质蛋白腱生蛋白C抑制肿瘤细胞的转移。
MiR-10b在转移性乳腺癌细胞系中作为致癌miRNA而被首次发现[7]。miR-10b水平与各种晚期癌症恶性程度相关。与早期原发性肿瘤相比,其在转移性肿瘤中表达上调。miR-10b直接靶向定位同源异型盒D10和Kruppel样因子4 。miR-21也是致癌因子,其能够抑制乳腺癌抑制基因的表达,从而促进肿瘤细胞生长、侵袭和转移。 miR-21的靶点,包括原肌球蛋白1α和程序性细胞坏死因子4[8]。MiR-21也靶向定位于10号染色体缺失的磷酸酶及张力蛋白同源物[9]以促进MCF-7型腺癌细胞生长[10]。
miR-155是的另一种致癌miRNA[11];其可以调节与细胞生长和存活有关的多重信号通路。人乳腺癌易感基因(breast cancer susceptibility gene,BRCA)1与DNA修复和细胞周期密切相关。miR-155靶向定位BRCA1,从而参与细胞修复以及影响细胞周期。miR-155还可以负调节与乳腺癌进展相关的其他基因(如细胞因子信号传导抑制因子1)。MiR-34a通常在乳腺癌中下调,这会使得沉默信息调节因子2相关酶1和B淋巴细胞瘤2基因的上调促进乳腺癌生长和存活[12-13]。miR-205经常在转移性乳腺癌中下调,miR-205的缺失使人表皮生长因子受体(human epidermal growth factor receptor,HER)3、血管内皮生长因子A、ZEB1和ZEB2蛋白上调,从而促进乳腺癌细胞生长和侵袭(图1)。

图1 mi-RNA在乳腺癌中的相互作用
注:↑表明基因或mi-RNA上调;↓代表基因或miRNA在乳腺癌组织中被下调;(-)为负性调节
细胞外基质和许多基质细胞,包括血管内皮细胞、淋巴管内皮细胞、癌相关成纤维细胞(cancer-associated fibroblasts,CAFs)、肿瘤相关巨噬细胞、间充质干细胞、免疫细胞、白细胞和脂肪细胞构成肿瘤微环境[14-15]。原发肿瘤有能力创造一种微环境与肿瘤发生部位相毗邻,被称为转移前肿瘤微环境。转移前微环境被发现有6个特征:能够促进以下过程、免疫抑制、炎症、血管生成/血管通透性、淋巴管生成、向器官性和重新排布[16]。转移细胞的miRNA能够促进转移性细胞向远处组织迁移、侵袭以及种植[17]。miRNAs也可以促进乳腺癌间质细胞的恶性转化。CAFs通过癌细胞之间的沟通,在癌症细胞转移过程中扮演重要角色[18]。CAFs由正常的成纤维细胞(normal fibroblasts,NFS)接受癌细胞分泌的生长因子和炎症因子的刺激后产生[19-20]。Baroni等[21]研究表明,人乳腺成纤维细胞可通过癌细胞分泌的miR-9获得类似CAFs的特性。miR-9能提高NFS的体外迁移率,并促进体内肿瘤生长。这一现象被瞬时转染miR-9的NFS证实。 Zhou等[22]报道MDA-MBB-231细胞分泌的miRNA与mcf10a细胞的分泌miRNA不同,可调节人微血管内皮细胞的迁移。miR-105在转移性乳腺癌细胞中过表达,并能够在内皮细胞中表达。此外, miR-105还可能通过下调胞质紧密连接蛋白1(zonula occludens-1,ZO-1)在一定程度上破坏人微血管内皮细胞单层膜的屏障功能。胞外体miR-105通过诱导血管通透性增强促进癌细胞的转移。转移的进展是通过上调miR-105在MCF-10A来源的致瘤细胞株的表达实现的。并且MDA-MB-231乳腺癌细胞中的miR-105抑制作用能够抑制癌细胞的转移并恢复血管的完整性。最终测量了miR-105和ZO-1的血清表达,表明miR-105与ZO-1的表达和转移有关,提示miR-105是转移性乳腺癌的一个潜在生物标志物[22]。Di Modica等[23]研究显示,miR-939来源于三阴性乳腺癌细胞,也可通过靶向定位于VE-钙黏素 (VE-cadherin,一种参与维持血管通透性的连接蛋白)破坏内皮的屏障功能。此外,miR-939与三阴性乳腺癌细胞患者预后差有关。Tominaga等[24]研究显示,来源于大脑转移癌细的miR-181c能够破坏血脑屏障,并建立了乳腺癌细胞株,即BMD2a和BMD2b,这些细胞系具有脑转移能力。在此基础上, He等[25]构建了一种血脑屏障体外模型,该模型由脑毛细血管内皮细胞、脑周细胞和星形胶质细胞组成。将BMD2a和BMD2b的胞外膜泡(extracellular vesicles,EVS)添加到上述模型中,观察肿瘤细胞对血脑屏障的高侵袭性。同样,EVS促进脑转移和紧密连接蛋白,即Claudin-5、occluin、ZO-1、N-cadherin(一种钙依赖性细胞间黏附蛋白)的定位,而且肌动蛋白在体内的定位出现明显的变化。miR-181c在脑转移癌细胞中表达过高。因此,得出结论:miR-181 c通过对上述蛋白的异常定位以及靶基因的下调而破坏血脑屏障。Fong等[26]研究了涉及转移前微环境的葡萄糖代谢的重编程能够促进糖代谢。结果表明,miR-122在体外可通过抑制糖代谢抑制基质细胞的增殖,而miR-122是乳腺癌细胞过度分泌的产物。M2型丙酮酸激酶为miR-122的靶点,其能够减轻miR-122对葡萄糖摄取的抑制作用。Hannafon等[27]确定一个具有抗血管生成和抗肿瘤作用天然化合物,名为二十二碳六烯酸,其能上调乳腺癌miRNAs-7a、miR-23b、miR-27a/b、miR-21、let-7和miR-320b,证实了其抗血管生成的特点。在内皮细胞中,过表达 miR-23b 和miR-320b能够通过靶向定位尿激酶型纤溶酶原激活因子、血管动蛋白样蛋白1、神经纤毛蛋白1抑制血管生成[27]。先前的研究表明,CAFs分泌趋化因子和细胞因子,如肝细胞生长因子和基质细胞衍生因子1,导致肿瘤转化,并增强了肿瘤微环境中的炎症和血管生成[28-30]。此外,在CAF外泌体中miR-21、miR-378 e和miR-143的表达水平高于NFS[31]。
3.1循环miRNA作为诊断生物标志物 Cuk等[32]探讨了2组乳腺癌组织中miR-127-3p、miR-148b、miR-376a、miR-376c、miR-409-3p、miR-652、miR-801的诊断潜力,观察到在乳腺癌患者中这些miRNA的表达上调,在乳腺癌Ⅰ期和Ⅱ期中miR-127-3p、miR-148b、miR-409-3p、miR-652、miR-801升高,表明它们可以用于早期诊断。另一项研究对乳腺癌患者和健康对照组血浆样品中的miRNA进行分析显示,43种miRNA的表达有差异,乳腺癌患者中miR-148b、miR-133a、miR-409-3p高表达[33]。最近的一项研究发现,与健康对照组比较,乳腺癌患者中miR-15a、miR-18a、miR-107、miR-133a、miR-139-5p、miR-143、miR-145、miR-365、miR-425表达上调[34]。另有研究者使用Taqman基因芯片技术研究早期乳腺癌患者和健康对照组中循环miR-484表达水平,发现在乳腺癌患者中更高[35]。在乳腺癌肿瘤患者和健康对照组血清样品中miRNA表达的全局分析显示,miR-1、miR-92a、miR-133a和miR-133b是乳腺癌血清中上调最明显的生物标志物[36]。Wang等[37]评估了乳腺癌患者循环miR-182的诊断潜力,与健康对照相比,乳腺癌患者循环miR-182的表达上调,并且在乳腺癌组织中高表达,表明其可以用作潜在的诊断生物标志物。尽管上述研究中,乳腺癌与miRNAs具有良好的一致性,但也有研究表明循环miRNA是非肿瘤源性的。Waters等[38]应用小鼠乳腺癌模型评估肿瘤进展期间循环miRNA表达的变化,发现小鼠乳腺癌模型中miR-138表达上调,并且其在乳腺癌患者血清中表达也上调。但乳腺癌组织本身并没有观察到miR-138表达水平的变化。
乳腺癌组织的miRNA表达谱显示,某些miRNA相关的表达模式与乳腺癌分子亚型、雌激素受体状态或其他病理特征相关[39]。因此,一些研究评估了循环miRNA在乳腺癌疾病分类中的潜在功能。Nassar等[40]研究发现,miR-155在孕激素受体阳性患者中高度表达。Wang等[41]观察到与正常组织相比,乳腺癌组织中miR-21、miR-106a和miR-155表达上调,而miR-126、miR-199a和miR-335表达下调,且miR-21、miR-126、miR-155、miR-199a和miR-335的表达水平与组织学肿瘤分级和性激素受体表达相关。
3.2循环miRNA作为疾病预后生物标志物 侵袭性乳腺癌患者的血清miR-202过表达与总体生存率降低呈正相关[42]。有研究尝试将miR-10b和miR-373循环水平作为检测乳腺癌淋巴结转移的生物标志物,与正常对照和没有转移的患者相比较,术前乳腺癌淋巴结转移患者血浆中miR-10b和miR-373表达水平较高[43]。另一项研究指出在原发性(M0)乳腺癌患者和转移性(M1)乳腺癌患者以及健康对照者中循环miR-34a表达上调与肿瘤分期相关,而miR-10b、miR-34a和miR-155表达上调与转移相关[44]。 Shaker等[45]发现miR-155和miR-205的表达与乳腺癌远处转移密切相关,与试验组乳腺癌相比,对早期乳腺癌和健康对照患者血清样品进行miRNA表达谱分析显示,miR-92a表达在组织和血清中早期下调,而miR-21的表达上调。此外,血清miR-92a和miR-21的表达水平与肿瘤大小和淋巴结转移存在相关性。miR-21-5p、miR-375、miR-205-5p和miR-194-5p表达上调和miR-382-5p、miR-376c-3p和miR-411-5p表达下调与乳腺癌复发相关[46]。另一项研究报道,循环miRNA表达与乳腺癌亚型有关联性,作者检测了M0和M1乳腺癌患者以及健康对照组血清中miR-10b、miR-17、miR-34a、miR-93、miR-155、miR-373的表达水平,结果显示M1期miR-17和miR-155的表达水平显着高于M0期,miR-373表达上调与原发性肿瘤的HER2阴性状态有相关性,而miR-17和miR-34a表达水平与孕激素受体或雌激素受体状态相关[47]。
3.3循环miRNA作为预测生物标志物 在一项研究中,在浸润性导管癌和术前辅助化疗的乳腺癌患者血清中miR-10b、miR-34a、miR-125b、miR-155表达水平高于正常对照组,其中在化疗不明显的乳腺癌患者中miR-125b表达水平更高,这表明miR-125b的表达与乳腺癌化疗耐药性之间可能存在相关性[48]。Wu等[49]对接受辅助化疗后局部手术切除肿瘤的Ⅱ~Ⅲ期乳腺癌患者进行了循环miRNA的测序,观察到miR-375表达下调和miR-122表达上调可用来区分复发和非复发患者,而在接受新辅助化疗的患者miR-375、miR-184、miR-1299和miR-196a表达上调,miR-381、miR-410和miR-1246表达下调,作者随后在第2组Ⅱ~Ⅲ期乳腺癌患者中发现了miR-122,并证实了循环miR-122表达上调与患者复发之间呈显着相关性,表明miR-122和miR-375在观察化疗疗效和预测乳腺癌复发方面有很大的应用前景。Sun等[50]检测乳腺癌患者与正常对照组血清中miR-155的表达水平,发现乳腺癌miR-155表达上调,且在术后和4个疗程化疗后血清中miR-155表达上调,表明miR-155有作为治疗反应指标的潜力。见表1。
表1 循环mi-RNA在乳腺癌中作为诊断、预后和预测的标记物
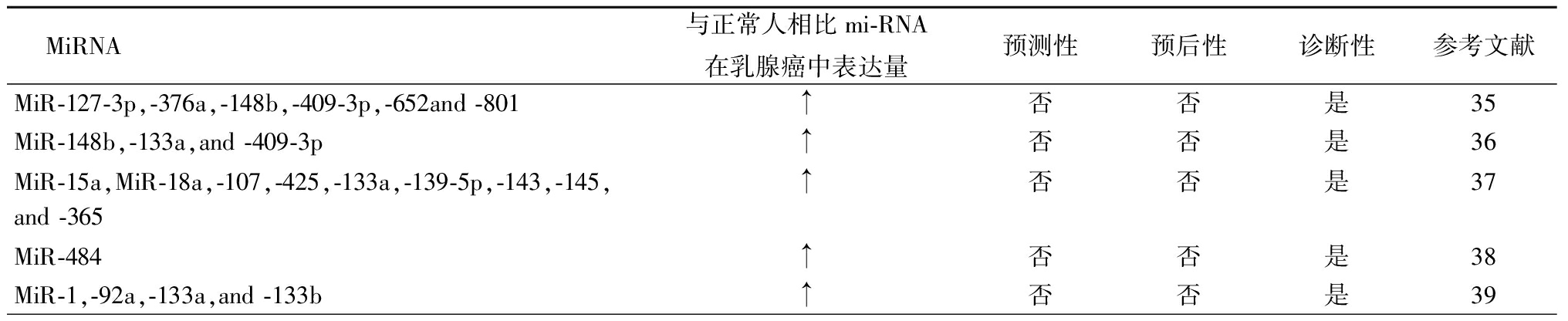
表1(续)
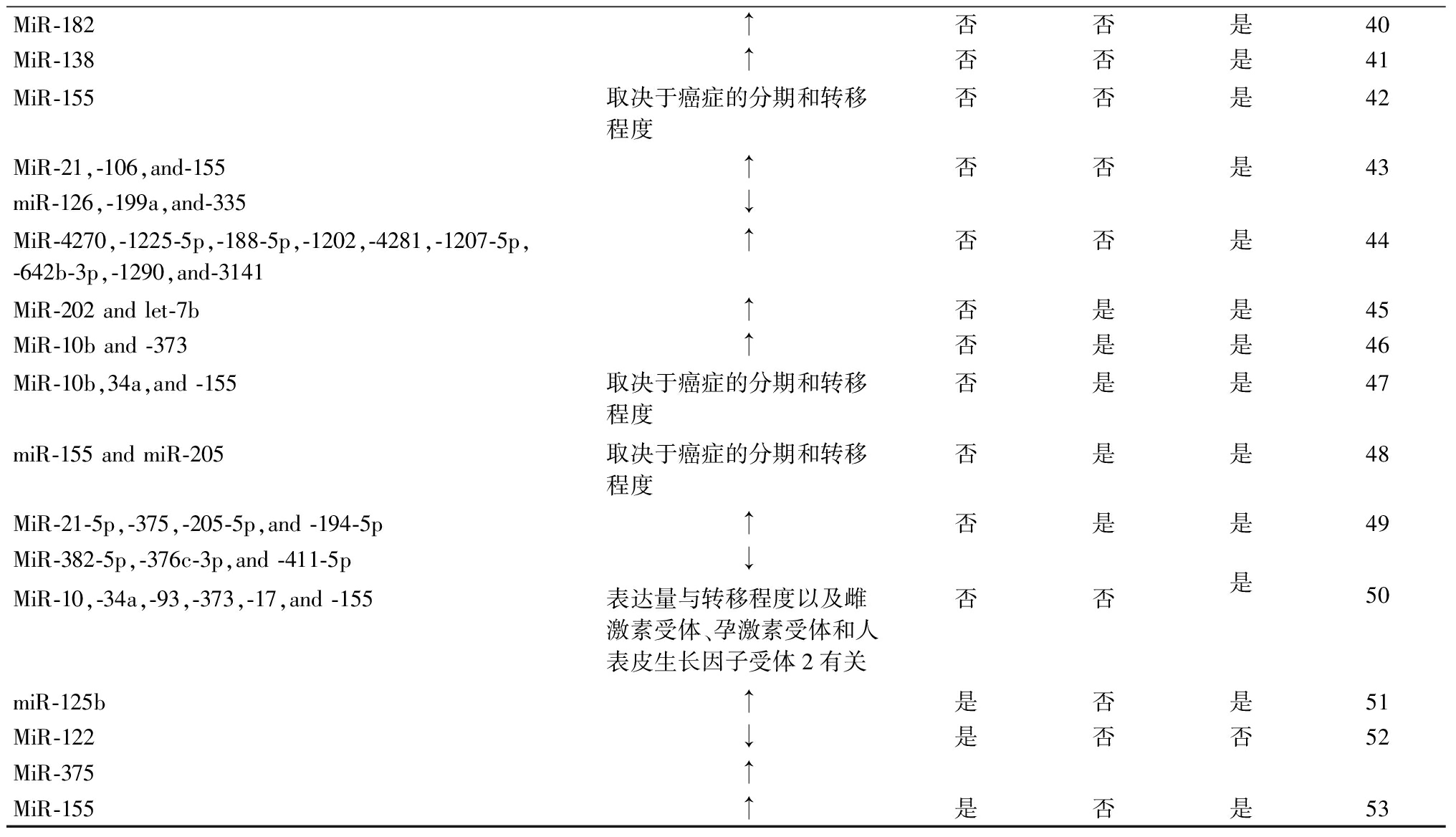
注:↑代表基因或mi-RNA表达上调;↓代表基因或mi-RNA表达下调
3.4miRNA作为乳腺癌生物标志物的局限性 建立一个准确可靠的用于乳腺癌诊断、预后和预测治疗反应的循环miRNA几乎每一步均具有挑战性[51]。第一,样品选择和处理,使用血清或血浆,以避免溶血造成的样品浪费,并采用标准化方案处理和收集。第二,癌症患者化疗前后miRNA水平波动[52-53],为了消除这个问题,可以按照用药前、用药时和用药后3个阶段收集血液样本。第三,miRNA测量平台的选择,基于qRT-PCR技术,虽然这种方法比其他方法更敏感且成本更低,但是主要的限制是仍存在检测盲区。第四,由于miRNA表达水平会随着生理和病理状态而改变,但目前缺乏可靠的措施标化miRNA表达。
循环miRNA在对乳腺癌患者诊断、预后以及疾病的管理方面具有很大的发展前景。目前临床上还没有有效的循环miRNA模板用于肿瘤学实践。这与患者选择不同、分离和测量循环miRNA的技术差异、miRNA低丰度、治疗效果和并发症、样本量不足、统计学分析不当等有关;与样品采集、测量方法和标准化相关的一些步骤,仍然需要进行标准和精简,而目前一些方法正在开发中,以增强检测的灵敏度和特异度,并改善miRNA的临床应用。
[参考文献]
[1] Siegel RL,Fedewa SA,Miller KD,et al. Cancer statistics for Hispanics/Latinos,2015[J]. CA Cancer J Clin,2012,65(6):457-480.
[2] 戴丹,徐燕丽.MicroRNA与恶性淋巴瘤相关性研究进展[J].河北医科大学学报,2009,30(10):1106-1108.
[3] Triboulet R,Pirouz M,Gregory RI. A Single Let-7 MicroRNA Bypasses LIN28-Mediated Repression[J]. Cell Rep,2015,13(2):260-266.
[4] Liang Z,Yi B,Zhao H,et al. Abstract 4790:Let-7 mediates sulindac inhibition of tumor cell transformation[J]. Cancer Res,2017,77(13 Suppl):4790.
[5] Chung VY,Tan TZ,Tan M,et al. GRHL2-miR-200-ZEB1 maintains the epithelial status of ovarian cancer through transcriptional regulation and histone modification[J]. Sci Rep,2016,6:19943.
[6] Jurmeister S,Baumann M,Balwierz A,et al. MicroRNA-200c represses migration and invasion of breast cancer cells by targeting actin-regulatory proteins FHOD1 and PPM1F[J]. Mol Cell Biol,2012,32(3):633-651.
[7] Zhang J,Yang J,Zhang X,et al. MicroRNA-10b expression in breast cancer and its clinical association.[J]. PLoS One,2018,13(2):e0192509.
[8] Pratheeshkumar P,Son YO,Divya SP,et al. Quercetin inhibits Cr(VI)-induced malignant cell transformation by targeting miR-21-PDCD4 signaling pathway[J]. Oncotarget,2016,8(32):52118-52131.
[9] Wang X,Hang Y,Liu J,et al. Anticancer effect of curcumin inhibits cell growth through miR-21/PTEN/Akt pathway in breast cancer cell[J]. Oncol Lett,2017,13(6):4825-4831.
[10] Li LQ,Li XL,Wang L,et al. Matrine inhibits breast cancer growth via miR-21/PTEN/Akt pathway in MCF-7 cells[J]. Cell Physiol Biochem,2012,30(3):631-641.
[11] Patel GK,Khan MA,Bhardwaj A,et al. Exosomes confer chemoresistance to pancreatic cancer cells by promoting ROS detoxification and miR-155-mediated suppression of key gemcitabine-metabolising enzyme,DCK[J]. Br J Cancer,2017,116(5):609-619.
[12] Li L,Yuan L,Luo J,et al. MiR-34a inhibits proliferation and migration of breast cancer through down-regulation of Bcl-2 and SIRT1[J]. Clin Exp Med,2013,13(2):109-117.
[13] Li L,Yuan L,Luo J,et al. MiR-34a inhibits proliferation and migration of breast cancer through down-regulation of Bcl-2 and SIRT1[J]. Clin Exp Med,2013,13(2):109-117.
[14] Turley SJ,Cremasco V,Astarita JL. Immunological hallmarks of stromal cells in the tumour microenvironment[J]. Nat Rev Immunol,2015,15(11):669-682.
[15] 高广周,张泽,郝英霞.肿瘤间隙液的研究进展[J].河北医科大学学报,2015,36(11):1359-1362.
[16] Liu Y,Cao X. Characteristics and significance of the pre-metastatic niche[J]. Cancer Cell,2016,30(5):668-681.
[17] Kalluri R. The biology and function of exosomes in cancer[J]. J Clin Invest,2016,126(4):1208-1215.
[18] Gascard P,Tlsty TD. Carcinoma-associated fibroblasts:orchestrating the composition of malignancy[J]. Genes Dev,2016,30(9):1002-1019.
[19] 蔡伟,康骅,刘晨,等.CAFs对乳腺癌细胞生物学特性的影响及机制探讨[J].现代肿瘤医学,2014,22(6):1249-1253.
[20] Swartz MA,Iida N,Roberts EW,et al. Tumor microenvironment complexity:emerging roles in cancer therapy[J]. Cancer Res,2012,72(10):2473-2480 .
[21] Baroni S,Romero-Cordoba S,Plantamura I,et al. Exosome-mediated delivery of miR-9 induces cancer-associated fibroblast-like properties in human breast fibroblasts[J]. Cell Death Dis,2016,7(7):e2312 .
[22] Zhou W,Fong MY,Min Y,et al. Cancer-secreted miR-105 destroys vascular endothelial barriers to promote metastasis[J]. Cancer Cell,2014,25(4):501-515 .
[23] Di Modica M,Regondi V,Sandri M,et al. Breast cancer-secreted miR-939 downregulates VE-cadherin and destroys the barrier function of endothelial monolayers[J]. Cancer Lett,2017,384:94-100 .
[24] Tominaga N,Kosaka N,Ono M,et al. Brain metastatic cancer cells release microRNA-181c-containing extracellular vesicles capable of destructing blood-brain barrier[J]. Nat Commun,2015,6:6716.
[25] He Y,Deng F,Yang S,et al. Exosomal microRNA:a novel biomarker for breast cancer[J]. Biomark Med,2018,12(2):177-188.
[26] Fong MY,Zhou W,Liu L,et al. Breast-cancer-secreted miR-122 reprograms glucose metabolism in premetastatic niche to promote metastasis[J]. Nat Cell Biol,2015,17(2):183-194.
[27] Hannafon BN,Carpenter KJ,Berry WL,et al. Exosome-mediated microRNA signaling from breast cancer cells is altered by the anti-angiogenesis agent docosahexaenoic acid (DHA)[J]. Mol Cancer,2015,14:133 .
[28] Konstorum A,Lowengrub JS. Activation of the HGF/c-Met axis in the tumor microenvironment:a multispecies model[J]. J Theor Biol,2018,439:86-99.
[29] Yang J,Lu Y,Lin YY,et al. Vascular mimicry formation is promoted by paracrine TGF-β and SDF1 of cancer-associated fibroblasts and inhibited by miR-101 in hepatocellular carcinoma[J]. Cancer Lett,2016,383(1):18-27.
[30] Sun Y,Wang R,Qiao M,et al. Cancer associated fibroblasts tailored tumor microenvironment of therapy resistance in gastrointestinal cancers[J]. J Cell Physiol,2018,233(9):6359-6369.
[31] Donnarumma E,Fiore D,Nappa M,et al. Cancer-associated fibroblasts release exosomal microRNAs that dictate an aggressive phenotype in breast cancer[J]. Oncotarget,2017,8(12):19592-19608 .
[32] Cuk K,Zucknick M,Madhavan D,et al. Plasma microRNA panel for minimally invasive detection of breast cancer[J]. PLoS One,2013,8(10):e76729.
[33] Shen J,Hu Q,Schrauder M,et al. Circulating miR-148b and miR-133a as biomarkers for breast cancer detection[J]. Oncotarget,2014,5(14):5284-5294.
[34] Kodahl AR,Lyng MB,Binder H,et al. Novel circulating microRNA signature as a potential non-invasive multi-marker test in ER-positive early-stage breast cancer:a case control study[J]. Mol Oncol,2014,8(5):874-883.
[35] Zearo S,Kim E,Zhu Y,et al. MicroRNA-484 is more highly expressed in serum of early breast cancer patients compared to healthy volunteers[J]. BMC Cancer,2014,14:200.
[36] Mangolini A,Ferracin M,Zanzi MV,et al. Diagnostic and prognostic microRNAs in the serum of breast cancer patients measured by droplet digital PCR[J]. Biomark Res,2015,3:12.
[37] Wang PY,Gong HT,Bao-Feng LI,et al. Higher expression of circulating miR-182 as a novel biomarker for breast cancer[J]. Oncol Lett,2013,6(6):1681-1686.
[38] Waters PS,Dwyer RM,Brougham C,et al.Impact of tumour epithelial subtype on circulating microRNAs in breast cancer patients[J]. PLoS One,2014,9(3):e90605.
[39] Xiao B,Zhang W,Chen L,et al. Analysis of the miRNA-mRNA-lncRNA network in human estrogen receptor-positive and estrogen receptor-negative breast cancer based on TCGA data[J]. Gene,2018,658:28-35.
[40] Nassar FJ,El Sabban M,Zgheib NK,et al.miRNA as potential biomarkers of breast cancer in the Lebanese population and in young women:a pilot study[J]. PLoS One,2014,9(9):e107566.
[41] Wang F,Zheng Z,Guo J,et al. Correlation and quantitation of microRNA aberrant expression in tissues and sera from patients with breast tumor[J]. Gynecol Oncol,2010,119(3):586-593.
[42] Joosse SA,Müller V,Steinbach B,et al. Circulating cell-free cancer-testis MAGE-A RNA,BORIS RNA,let-7b and miR-202 in the blood of patients with breast cancer and benign breast diseases[J]. Br J Cancer,2014,111(5):909-917.
[43] Chen W,Cai F,Zhang B,et al. The level of circulating miRNA-10b and miRNA-373 in detecting lymph node metastasis of breast cancer:potential biomarkers[J]. Tumour Biol,2013,34(1):455-462.
[44] Roth C,Rack B,Müller V,et al. Circulating microRNAs as blood-based markers for patients with primary and metastatic breast cancer[J]. Breast Cancer Res,2010,12(6):R90.
[45] Shaker O,Maher M,Nassar Y,et al. Role of microRNAs-29b-2,-155,-197 and -205 as diagnostic biomarkers in serum of breast cancer females[J]. Gene,2015,560(1):77-82.
[46] Huo D,Clayton WM,Yoshimatsu TF,et al. Identification of a circulating MicroRNA signature to distinguish recurrence in breast cancer patients[J]. Oncotarget,2016,7(34):55231-55248.
[47] Eichelser C,Flesch-Janys D,Chang-Claude J,et al. Deregulated serum concentrations of circulating cell-free MicroRNAs miR-17,miR-34a,miR-155,and miR-373 in human breast cancer development and progression[J].Clin Chem,2013,59(10):1489-1496.
[48] Wang H,Tan G,Dong L,et al. Circulating MiR-125b as a marker predicting chemoresistance in breast cancer[J]. PLoS One,2012,7(4):e34210.
[49] Wu X,Somlo G,Yu Y,et al. De novo sequencing of circulating miRNAs identifies novel markers predicting clinical outcome of locally advanced breast cancer[J]. J Trans Med,2012,10:42.
[50] Sun Y,Wang M,Lin G,et al. Serum microRNA-155 as a potential biomarker to track disease in breast cancer[J]. PLoS One,2012,7(10):e47003.
[51] Witwer KW. Circulating microRNA biomarker studies:pitfalls and potential solutions[J]. Clin Chem,2015,61(1):56-63.
[52] Diener Y,Walenda T,Jost E,et al. MicroRNA expression profiles of serum from patients before and after chemotherapy[J]. Genom Data,2015,6:125-127.
[53] Ponomaryova AA,Morozkin ES,Rykova EY,et al. Dynamic changes in circulating miRNA levels in response to antitumor therapy of lung cancer[J]. Exp Lung Res,2016,42(2):95-102.
[中图分类号]R737.9
[文献标志码]A
[文章编号]1007-3205(2018)09-1100-07
[收稿日期]2018-03-12;
[修回日期]2018-04-13
[基金项目]国家自然科学基金项目(81372150)
[作者简介]黄耀孟(1992-),男,河北河间人,河北医科大学医学硕士研究生,从事医学生物化学与分子生物学研究。
*通讯作者。E-mail:lbh58@hebmu.edu.cn
(本文编辑:赵丽洁)