脊髓性肌萎缩症(spinal muscular atrophy,SMA)是一组脊髓前角及脑干运动神经元变性、丢失,导致的以缓慢进行性加重、肢体近端对称性肌无力、肌萎缩为主要临床表现的遗传性骨骼肌疾病,呈常染色体隐性遗传[1]。95%的SMA由运动神经元生存基因(survival motor neuron,SMN)拷贝缺失致病,5%SMN点突变致病。婴幼儿、青少年为主要发病人群。群体发病率1/6 000~1/10 000,携带频率1/35~1/42,无明显地域及人口种族差异性[2-3]。SMA存在诊断分型难,儿童发病、致死率高、药物治疗费用极高且治疗手段有限等科学问题。全美医学遗传学会推荐将SMA携带者产前筛查纳入常规孕前检查项目[4]。本研究以分子生物学方法对4 568例样本进行SMA携带者筛查,并建立SMA分子生物学诊断、鉴别诊断平台同时完善该病产前策略,报告如下。
1 资 料 与 方 法
1.1 一般资料 选取2017年1月—2018年12月因孕前检查于石家庄市第四医院产前诊断中心就诊的患者4 568例,女性2 480例,男性2 088例,对检出的携带者配偶126例及夫妇双方均为携带者25对孕期胎儿行针对性检测。
本研究经医院伦理委员会审批通过,所有患者均知情同意且签署知情同意书。
1.2 介入性产前诊断 孕妇取仰卧位,常规消毒铺巾,超声定位穿刺部位。孕12周孕妇13例行绒毛穿刺术,由专人超声引导下经腹部或经阴道抽取妊娠绒毛组织25 mg;余孕18周后行羊水穿刺术,由专人经腹部抽取羊水30 mL。
1.3 分子生物学方法
1.3.1 DNA提取、聚合酶链反应(polymerase chain reaction,PCR)扩增 抽取3 mL外周血,天根DNA提取试剂盒获得基因组DNA。
羊水细胞培养,7 d后以生理盐水反复冲洗5~6次贴壁羊水细胞并进行细胞收集,应用天根DNA细胞基因组试剂盒提取胎儿羊水细胞基因组DNA。NanoDrop1000微量核苷酸蛋白多功能酶标仪(美国Gene公司)定量分析(OD260/OD280比值1.8~2.0),-80 ℃保存余DNA样本。Prime 6软件设计引物,覆盖SMN1/KRIT1/CYBB全长,包括目的基因片段各个外显子及与内含子交界区,Pubmed blast比对引物(表1)。KRIT1/CYBB为内参基因。PCR扩增参数:95 ℃ 10 min热启动预变性,95 ℃ 30 s,54 ℃30 s,72 ℃ 60 s,共25个循环,72℃充分延伸10 min。2%琼脂糖凝胶电泳,100 V,30 min。
1.3.2 Denaturing high performance liquid chromatography(DHPLC)分析 样本完成PCR扩增进行DHPLC检测。5 μL PCR反应样本上机(美国环球基因公司),选择类型DS Multiple Fragment,温度50 ℃,上样、洗脱。应用仪器自带软件计算SMN1/KRIT1比值,SMN1/KRIT1等于0纯合缺失,杂合缺失或携带为0.5,正常人为1。以SMN1/KRIT1比值作为校正系数,所有比值按照校正系数进行比对后得出数据。
1.3.3 Sanger法测序 美国ABI 3730XL(Applied Biosystems,Foster City,CA)自动测序仪测序。Sequencher v4.1(Gene Codes,Ann Arbor,MI,USA)比对分析,致病变异位点进行家系成员靶向检测分析。分析变异序列与疾病表型有无共分离现象,进行基因变异位点定位、明确氨基酸变化及功能改变。
2 结 果
2.1 DHPLC检测结果 研究以DHPLC方法对入选4 568例样本进行SMN 1外显子进行拷贝缺失检测,2例SMN1外显子7/8纯合缺失(SMA患者),父母双方验证SMN1外显子7/8杂合缺失,拷贝数“1”(典型SMA携带者),余样本中未见SMN1外显子7及外显子8纯合缺失;共计检测到携带者126例,携带频率1/36,其中SMN1外显子7/8杂合缺失84例,拷贝数均为“1”,仅SMN1外显子7拷贝数“1”31例,单独SMN1外显子8拷贝数“1”11例,致病基因携带频率1/40。联系并告知上述携带者配偶并在其知情同意基础上对其抽取外周血进行SMN 1外显子拷贝变异检测,25例配偶样本证实为SMN1外显子7/8杂合缺失,即拷贝数均为“1”,致病携带率1/50(图1)。
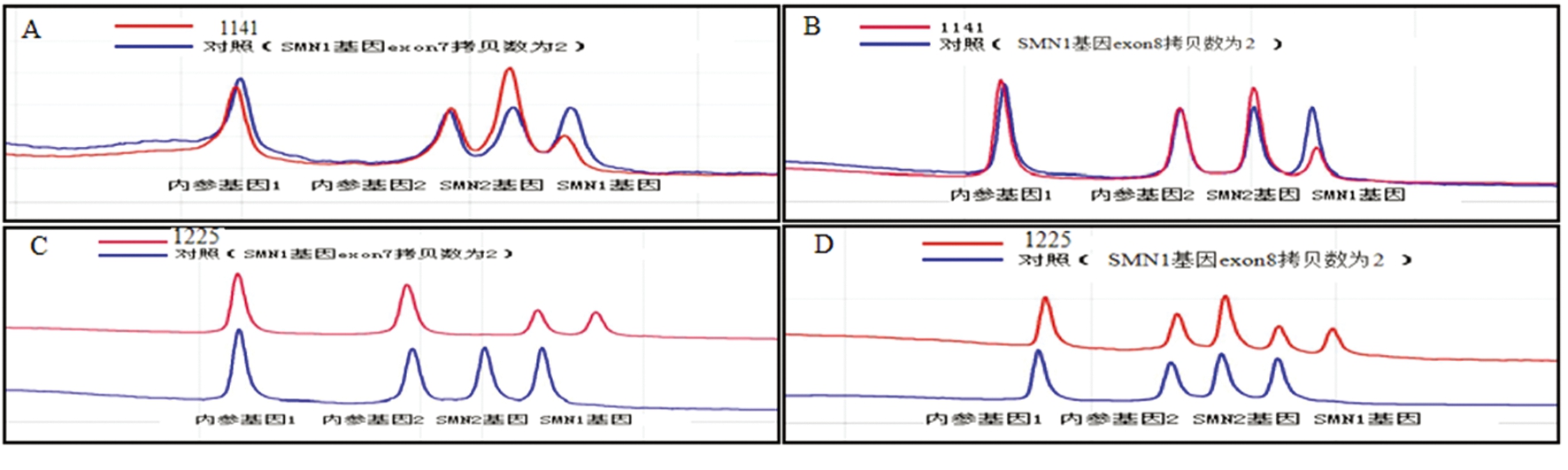
图1 DHPLC检测SMN1缺失检测
A.表明SMA携带者SMN17号外显子杂合缺失,拷贝数为“1”;B.表明SMA携带者SMN1 8号外显子杂合缺失,拷贝数为“1”;C.表明SMA患者SMN1 7号外显子杂合缺失,拷贝数为“0”;D.表明SMA患者SMN1 8号外显子杂合缺失,拷贝数为“0”
Figure 1 SMN1 deletion was detected by DHPLC
2.2 Sanger测序检测结果 研究对4 568例样本进行SMN1点突变变异检测,明确1例SMN1外显子7/8杂合缺失携带者拷贝数为“1”样本同时携带p.Q164X点突变变异位点,临床诊断为“1+1型”SMA患者,告知该样本父母检测结果并知情同意前提下对父母行SMN1缺失及点突变检测,证实其母为SMN1外显子7及外显子8杂合缺失携带者,其父亲为p.Q164X点突变携带者(图2)。余样本(含拷贝缺失携带者配偶)未检测出致病意义明确变异位点。
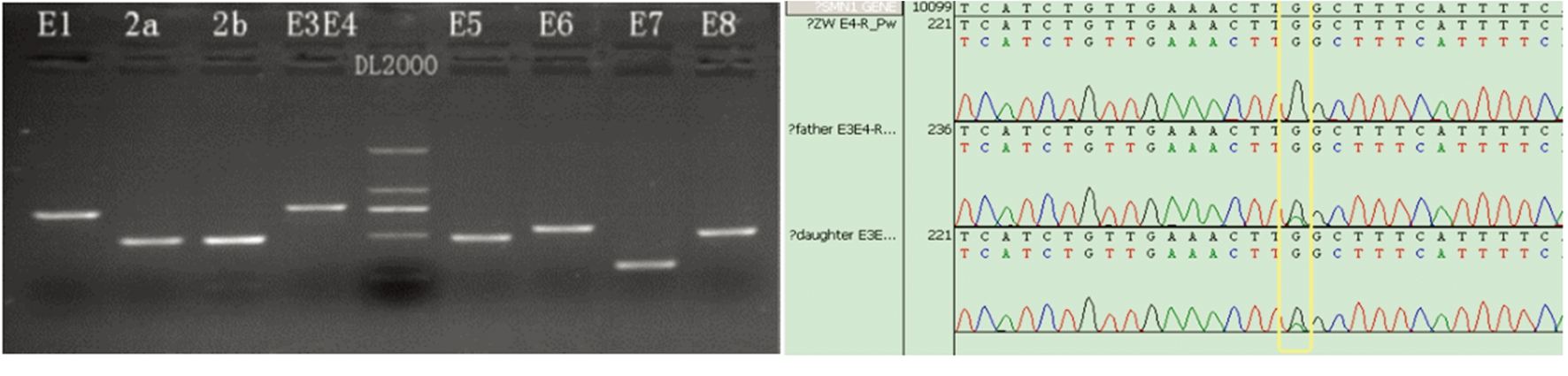
图2 SMN1 Sanger测序分析结果
左:DAN扩增样本琼脂糖凝胶电泳;右:p.Q164X(C>T)正反向测序形成终止密码子TAA
Figure 2 Results of SMN1 Sanger sequencing analysis
2.3 SMA携带者高危胎儿产前诊断 研究对上述SMA携带者中25对夫妇(含点突变夫妇)胎儿于孕18周行羊水穿刺,并应用DHPLC及Sanger测序技术进行SMN1拷贝数及点突变变异检测。结果显示SMN1外显子7/8纯合缺失(SMA患者)胎儿3例;SMN1外显子7/8杂合缺失(SMA携带者)胎儿1例。余胎儿检测未见SMN1拷贝数异常(图3)。Sanger测序1例胎儿携带p.Q164X点突变变异位点,余样本未见SMN异常。
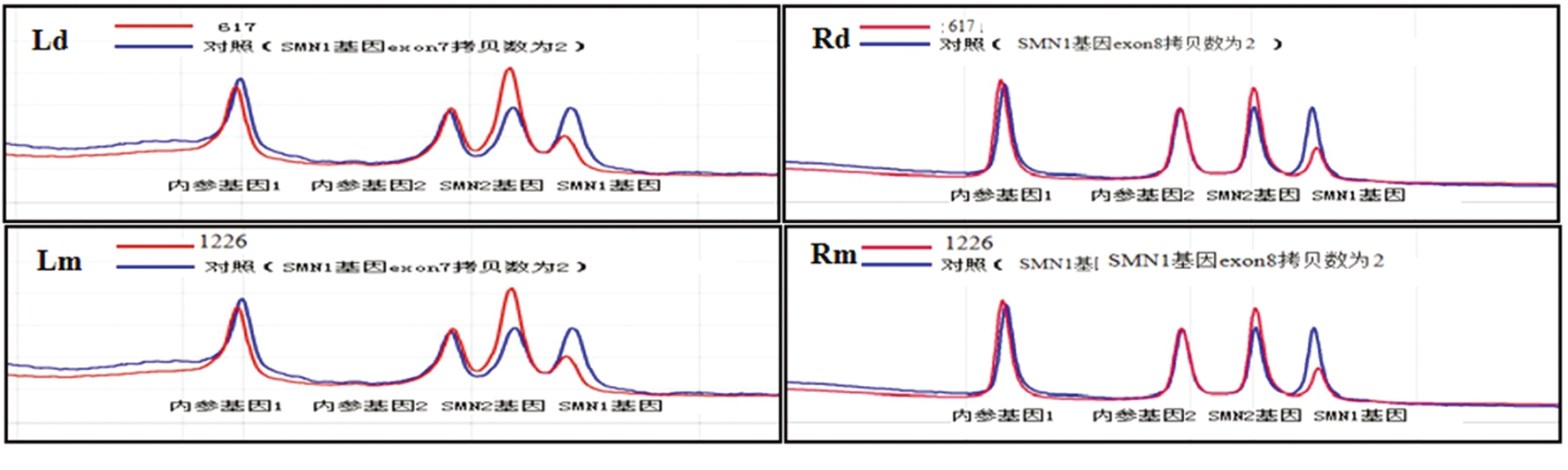
图3 DHPLC对SMA携带者高危胎儿SMN1检测分析
Ld.被检测孩子样本SMN1 7号外显子杂合缺失;Rd.被检测孩子样本SMN1 8号外显子杂合缺失;Lm.被检测母亲样本SMN1 7号外显子杂合缺失;Rm.被检测母亲样本SMN1 8号外显子杂合缺失
Figure 3 Detection and analysis of SMN1 in high risk fetuses with SMA carriers by DHPLC
3 讨 论
SMA是仅次于杜兴型肌营养不良(Duchenne muscular dystrophy,DMD)常见的神经骨骼肌疾病,也是目前已知除囊性纤维变外最常见致死性极高的常染色体隐性遗传病。美国食品药品监督管理局于2019年批准首个治疗SMA基因治疗药物Zolgensma(onasemnogene abeparvovec-xioi)上市并应用于临床,但1剂用药212万美元的高昂费用且仅针对2岁以下儿童用药范围限制了其短期内临床普及进度[5-6]。因此,临床对先证者早期诊断及携带者早期筛查有重要意义。既往研究证实[7-8]:①染色体5q11.2-q13.3区域SMN变异导致脊髓前角细胞与脑干内运动核进行性变性为其发病机制;②SMA是婴儿期最常见致死性遗传病;③肌电图、肌酶和肌肉活检诊断特异度不高,分子生物学诊断检测“金标准”;④“SMA 0型”被报道,遗传学超声可见宫内胎动明显减少,如不及时采取措施,1岁内即死亡。上述特征为SMA临床诊断、分型、治疗、预防提出了难题。分子生物学技术逐渐成为不可取代的诊断、分型SMA“金标准”。悉数SMA分子检测手段,由最初的聚合酶链-单链构象多态性分析、限制性片段长度多态性分析、DHPLC、PCR技术、到多重连接依赖式探针扩增(multiplex ligation dependent probe amplification,MLPA)[9],技术手段各有优劣,寻找精准、有效、经济的分子检测手段成为临床医生关注的焦点。
我国学者对338例SMA疑似患儿行PCR-RFLP分析中发现267例为纯合缺失,另外88例中17例符合SMA国际诊断标准,经过DHPLC验证为杂合缺失;同时有研究证实在对85例临床疑似SMA患儿进行PCR-RFLP分析57例考虑纯合缺失,另外19例阴性患儿后期随访过程中发现并不符合SMA诊断。由此我们不难发现单独依靠PCR-RFLP容易造成SMA误诊、漏诊,同时单将限制性片段长度多态性分析用于产前诊断无法准确判断母源污染而存在假阴性率。与PCR-RFLP技术相比,DHPLC是一类单核苷酸多态性研究中常见技术,能明确患儿及其亲属SMN缺失、杂合缺失情况并解决酶切不完全、限制酶失效等问题,此外还具备成本低、检测快速、通量高等优势[10]。本研究基于SMA流行病学报道及发病特征,选择DHPLC进行检测,同时结合单基因靶向测序技术进行“点突变”致病位点检测筛查,二者有效结合,防“漏筛”同时有效降低消耗成本。
研究结果中128例SMN 1拷贝异常样本中2例明确SMA,余126例携带者,携带频率1/36,与1/35~1/168不同人群携带波动频率比较[11],我省地区携带频率与欧美国家及我国南方人群(含台湾地区)遗传流行病学研究报告携带频率1/25~1/50较为一致。众所周知,SMN1中外显子7决定SMA临床表型及致病意义,SMA患者SMN1一般表现为外显子7、8同时缺失或仅外显子7纯合缺失。SMN1中外显子8缺失与否并不能导致SMN1编码蛋白结构及功能的改变,而且鲜有SMN1外显子8缺失致病报道[12],因此SMN1外显子7缺失被公认为SMA致病原因。但SMA患者SMN1与SMN2转化过程中会引起外显子8的多态性改变,因此8号外显子缺失通常作为后期随访及观察组,也是SMN1基因缺失中不可缺少的分析位点[13]。
测序技术是SMN1缺失检测有效补充,既可以对患者进行疾病热点突变位点-临床表型分析也可以靶向判断碱基改变,对新发变异突变位点与遗传病间关系研究起着不可替代的作用。本研究1例“1+1”型SMA样本Sanger测序结果显示位于6号外显子无义突变位点p.Q164X为亚洲人群首报且经蛋白水平检测致病意义明确。迄今,已发现的点突变致病位点约30余个且多分布于3号/6号外显子[13],本研究证实6号外显子为“热点突变”占据地。这一检测结果填补了中国SMA点突变基因数据库。该SMA患儿临床发病晚、症状轻,仅表现双下肢肌力减退及远端肌轻度萎缩。Wu等[14]指出SMA患者临床表型严重程度除与SMN1与SMN2“剂量补偿”有关,“1+1型”患者临床表型与“点突变”类型密切相关,如无义突变致病较轻微,而严重的1型患者多与移码突变相关。我们对样本亲缘验证示SMN1缺失变异源自其母,p.Q164X源自其父。该验证结果与约2%新发点突变变异位点中大部分源于父系报道一致[15]。因此,点突变测序技术是SMA分型诊断必不可少的检测途径。
基因筛查与基因诊断最大的区别就是应用人群及检测目标的差异,但却有着相同的遗传学基础及极为相似的检测手段,因此人群携带者筛查最终落实的实际意义在于指导高危家庭的遗传咨询、妊娠及如何规避缺陷患儿出生。SMA作为AR遗传病符合孟德尔遗传定律,即如夫妇双方均为携带者,子代表现为25%发病,50%携带,25%正常[16]。本研究对前期明确25对SMA携带者夫妇胎儿行介入性产前诊断SMA占12%,携带率达8%。该数据证实SMA产前筛查及产前诊断极为重要,实际临床工作中医疗环境及医生认识问题使部分医生忽略SMA携带者筛查指导及产前策略分析。
目前已知SMA患儿防治技术主要有:①SMA无创产前诊断,通过检测孕妇外周血中胎儿游离DNA及对孕妇中胎儿有核细胞进行富集后而进行的胎儿遗传学诊断;②通过胚胎植入前遗传学诊断技术对胚胎基因组变异进行明确诊断并剔除不良胚胎;③基于携带者筛查基础上于孕中期行胎儿产前诊断[17-19]。显然,产前诊断是最精准、经济、有效的手段。那么在我国什么样的人群适合SMA携带者筛查呢,当然,对于各项医疗保障制度完善的地区我们提倡“谱筛”,指导携带者夫妇遗传咨询及生育并选择产前诊断最佳时机;对于无条件执行谱筛的医疗条件下我们建议对下列人群靶向进行SMA携带者筛查:①既往生育过SMA患儿的夫妇;②遗传学超声提示胎儿胎动少,伴有室间隔缺损及四肢姿势固定、关节挛缩的影像学表现;③夫妇双方家系中均有SMA发病病史的;④夫妇双方一方携带者或患者欲生育的;⑤先证者经基因诊断明确的高危产妇的。Chen等[20]对SMA筛查评估证实SMA患儿从2003年90.62%逐渐下降到2014年的20.83%,且检测结果益处大于弊处。由此可见,精准筛查、严格把控筛查指征及科学严谨遗传指导可有效降低SMA缺陷患儿的出生,造福人类。
综上所述,SMA携带者筛查可谓是预防措施的“双刃剑”。SMA精准的筛查可以降低SMA患儿的出生,提供有效的遗传咨询信息,如预测不同人群地区种族的携带及发病频率,更可作为遗传评估手段用于辅助生殖,使家庭再次生育健康孩子的愿望得以实现。我国学者Tisdale等[17]早期曾建立了对先证者进行拷贝及点突变检一套完整检测系统,指出建立完整的SMA携带者筛查流程在该病预防及遗传咨询中有重要作用,因此我们认为完善的携带者筛查分析进行拷贝缺失、点突变检测分析固然重要,但对于种族及地区间的差异分析也是评估携带者筛查的重要因素。本研究通过上述人群研究总结出河北地区人口SMA人群携带频率及发病频率。明确携带者基础上,进行规范遗传咨询及生育指导。
[1] Polido GJ,de Miranda MMV,Carvas N,et al. Cognitive performance of children with spinal muscular atrophy:a systematic review[J]. Dement Neuropsychol,2019,13(4):436-443.
[2] Kou HS,Wang CC. Molecular inversion probes equipped with discontinuous rolling cycle amplification for targeting nucleotide variants:determining SMN1 and SMN2 genes in diagnosis of spinal muscular atrophy[J]. Anal Chim Acta,2017,977:65-73.
[3] Friedman LR,Kwon JM. Spinal muscular atrophy:past,present,and future[J]. Neoreviews,2019,20(8):437-451.
[4] Moultrie RR,Kish-Doto J,Peay H,et al. A review on spinal muscular atrophy:a wareness,knowledge and attitudes[J]. J Genetic Couns,2016,25(5):892-900.
[5] Ross LF,Kwon JM. Spinal muscular atrophy:past,present,and future[J]. Neoreviews,2019,20(8):e437-e451.
[6] Tisdale S,Pellizzoni L. Disease mechanisms and therapeutic approaches in spinal muscular atrophy[J]. J Neurosci,2015,35(23):8691-8700.
[7] Boardman FK,Sadler C,Young PJ,et al. Newborn genetic screening for spinal muscular atrophy in the UK:the views of the general population[J]. Mol Genet Genomic Med,2018,6(1):99-108.
[8] Zhang J,Wang Y ,Ma D,et al. Carrier screening and prenatal diagnosis for spinal muscular atrophy in 13,069 chinese pregnant women[J]. J Mol Diagn,2020,22(6):817-822.
[9] Matias L,Amber N,Vamshi K. Spinal muscular atrophy diagnosed by newborn screening[J]. Pediatr Neurol Briefs,2019,33(5):447-456.
[10] Prior TW,Nagan N. Spinal muscular atrophy:overview of molecular diagnostic approaches[J]. Curr Protoc Hum Genet,2016,88:9.27.1-9.27.13.
[11] Ar Rochmah M,Shima A,Harahap NIF,et al. Gender effects on the clinical phenotype in japanese patients with spinal muscular atrophy[J]. Kobe J Med Sci,2017,63(2):E41-E44.
[12] Cao YY,Zhang WH,Qu YJ,et al. Diagnosis of spinal muscular atrophy:a simple method for quantifying the relative amount of survival motor neuron gene 1/2 using sanger DNA sequencing[J]. Chin Med J(Engl),2018,131(24):2921-2929.
[13] Calucho M,Bernal S,Alías L,et al. Correlation between SMA type and SMN2 copy number revisited:an analysis of 625 unrelated Spanish patients and a compilation of 2834 reported cases[J]. Neuromuscul Disord,2018,28(3):208-215.
[14] Wu S,Li YL,Cheng NY,et al. c.835-5T>G Variant in SMN1 gene causes transcript exclusion of exon 7 and spinal muscular atrophy[J]. J Mol Neurosci,2018,65(2):196-202.
[15] Al-Zaidy SA,Mendell JR. From clinical trials to clinical practice:practical considerations for gene replacement therapy in SMA type 1[J]. Pediatr Neurol,2019,100:3-11.
[16] Parente V,Corti S. Advances in spinal muscular atrophy therapeutics[J]. Ther Adv Neurol Disord,2018,11(5):1-13.
[17] Tisdale S,Pellizzoni L. Disease mechanisms and therapeutic approaches in spinal muscular atrophy[J]. J Neurosci,2015,35(23):8691-8700.
[18] Vidal-Folch N,Gavrilov D,Raymond K,et al. Multiplex droplet digital PCR method applicable to newborn screening,carrier status,and assessment of spinal muscular atrophy[J]. Clin Chem,2018,64(12):1753-1761.
[19] Feng Y,Ge X,Meng L,et al. The next generation of population-based spinal muscular atrophy carrier screening:comprehensive pan-ethnic SMN1 copy-number and sequence variant analysis by massively parallel sequencing[J]. Genet Med,2017,19(8):936-944.
[20] Chen YC,Chang JG,Liu TY. Securinine enhances SMN2 exon 7 inclusion in spinal muscular atrophy cells[J]. Biomed Pharmacother,2017,88:708-714.