乳腺癌是女性最常见的恶性肿瘤,其发病率呈逐年上升的趋势,研究显示,2018年全球乳腺癌新发病例约208万,死亡63万[1];在我国2015年乳腺癌发病率占女性肿瘤的15%,病死率达69.5/1 000人[2]。治疗后复发转移仍是乳腺癌死亡的主要原因。随着对乳腺癌研究的深入,免疫治疗和靶向治疗的发展,乳腺癌的病死率明显下降,但复发和转移仍是目前临床治疗中急需解决问题之一。因此深入研究乳腺癌发生发展分子生物学机制,寻找乳腺癌早期诊断及预后评估的分子生物学标志物至关重要。随着基因芯片技术、高通量测序和精准医学的快速发展,研究发现长非编码RNA(long noncoding RNA,LncRNA)在肿瘤的发生发展中发挥着重要作用,对肿瘤的早期诊断及预后评估具有重要的临床应用价值[3]。并且在肿瘤中LncRNA对免疫细胞浸润、抗原识别、肿瘤细胞清除等免疫应答中发挥十分重要的调控作用[4]。既往研究显示多种LncRNA如SNHG12、CCHE1、LINC01833、LINC00536和LINC02725的表达与乳腺癌发生发展及预后密切相关,可作为乳腺癌预后标志物[5-7]。最近也有研究报道在乳腺癌中LncRNA表达谱预测模型的构建,并证实与乳腺癌预后显著相关[6]。本研究通过下载癌症基因组图谱(the cancer genome atlas,TCGA)中1 041例乳腺癌患者转录组测序数据和临床预后信息,探讨乳腺癌中免疫相关LncRNA表达与肿瘤预后的关系,通过筛选乳腺癌患者预后相关免疫LncRNA,构建准确预测乳腺癌患者预后的预后风险模型,为乳腺癌患者预后评估提供指导。
1 材料与方法
1.1 材料来源 本研究从TCGA 数据库(https://cancergenome.nih.gov/)下载1 041例乳腺癌组织样本转录组数据,所有样本均包含完整的高通量测序counts数据,同时下载乳腺癌患者的临床数据。包括:年龄、TNM分期、肿瘤分期、淋巴结转移、远处转移、总生存时间和生存状态;排除生存期<30 d、临床分期不明确以及预后信息不详的乳腺癌患者。数据下载截止日期到2020年10月11日TCGA收录的乳腺癌转录组数据。
1.2 获取LncRNA表达矩阵 从TCGA 数据库(https://cancergenome.nih.gov/)下载1 041例乳腺癌转录组数据相应的manifest和metadata文件,使用Perl软件及语言脚本提取乳腺癌原始的counts数据,从Ensembl数据库(https://asia.ensembl.org/index.html)下载Homo-sapiens.GRCh38.95.chr.gtf.gz文件,进行转录组数据表达谱id转换,得到gene symbol乳腺癌基因表达谱矩阵,使用Perl语言脚本提取其中的乳腺癌LncRNA的表达谱矩阵。
1.3 获取乳腺癌免疫相关LncRNA 从GSEA数据库(https://www.gsea-msigdb.org/gsea/index.jsp)下载免疫相关基因集(Immune response M19817,Immune system process M13664),使用Perl语言脚本获取乳腺癌免疫相关基因,进一步通过R语言的“limma”包使用共表达方法,以相关系数Cor>0.6和P<0.001提取乳腺癌免疫相关LncRNA[8]。
1.4 获取乳腺癌预后相关LncRNA构建预后风险模型 使用R软件中的survival包对1 041例乳腺癌免疫相关LncRNA表达矩阵进行单因素Cox回归筛选免疫预后相关LncRNA,进一步纳入多因素Cox回归分析,根据Akaike Information Criterion(AIC)确定最佳LncRNA建立多基因预后风险模型,基于所选择的LncRNA基因表达量乘以多因素Cox回归系数之和计算每例患者的风险值(risk score),根据中位风险值将乳腺癌患者分为低风险组和高风险组。
1.5 统计学方法 应用R软件(4.0.4版),采用Kaplan-Meier生存分析对两个亚组预后差异进行检验,并绘制生存曲线,同时绘制生存预测的ROC曲线,并计算曲线下面积AUC值对预后风险模型进行验证。同时结合临床病理信息及每例患者的risk score值进行单因素和多因素Cox回归分析,并绘制森林图,对预后风险模型进行验证。P<0.05为差异有统计学意义。
2 结 果
2.1 病例特征 本研究共纳入1 041例乳腺癌患者,年龄26~90岁,平均(58.19±13.20)岁。临床病理特征见表1。
表1 1 041例乳腺癌患者临床病理特征
Table 1 Clinicopathological characteristics of 1 041
breast cancer patients

临床指标例数(%)年龄 <60岁559(53.70) ≥60岁482(46.30)临床分期 Ⅰ172(16.52) Ⅱ589(56.58) Ⅲ237(22.77) Ⅳ19(1.83) 不确定24(2.30)T分期 T1269(25.84) T2604(58.02) T3129(12.39) T436(3.46) 不确定3(0.29)M分期 M0866(83.19) M121(2.02) 不确定154(14.79)N分期 N0480(46.11) N1348(33.43) N2118(11.34) N375(7.20) 不确定20(1.92)
2.2 免疫相关LncRNA与乳腺癌患者预后关系 从Ensembl数据库下载Homo-sapiens.GRCh38.95.chr.gtf.gz文件,进行id转换,得到gene symbol乳腺癌基因表达谱矩阵,使用Perl语言脚本提取其中的乳腺癌14 142个LncRNA的表达谱矩阵。从GSEA数据库下载免疫相关基因集Immune response M19817和Immune system process M13664,使R语言的“limma”包以共表达方法,设定相关系数Cor>0.6和P<0.001提取乳腺癌免疫相关LncRNA共644个。使用R软件中的“survival”包对644个乳腺癌免疫相关LncRNA表达矩阵进行单因素Cox回归筛选免疫预后相关LncRNA 14个。见表2。
表2 单因素Cox回归分析筛选乳腺癌免疫预后相关LncRNA
Table 2 Univariate Cox regression analysis to screen breast cancer immune prognosis-related LncRNA
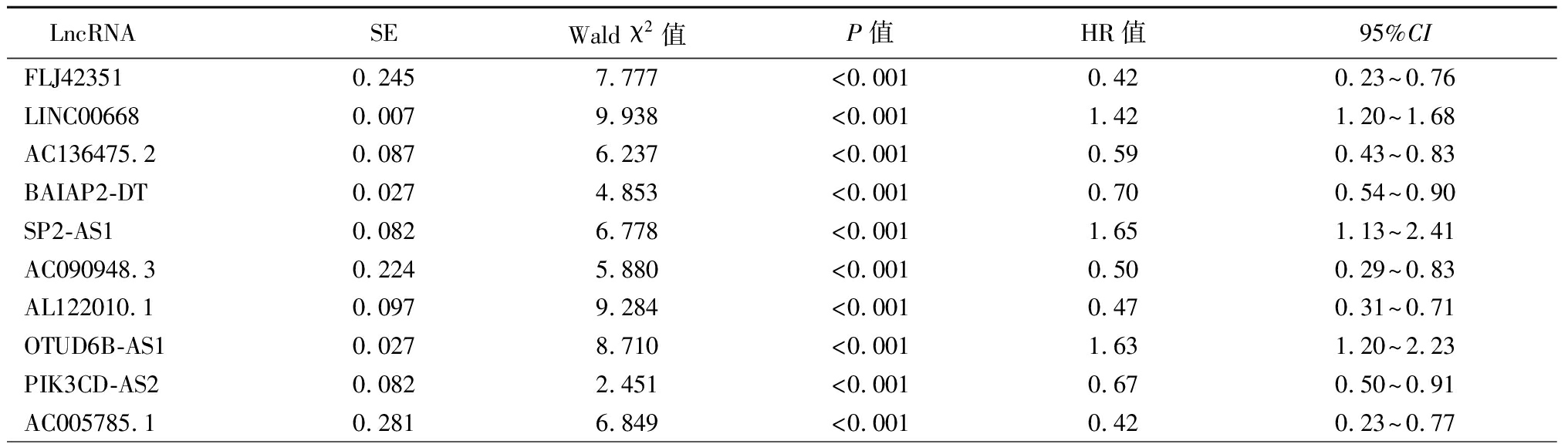
LncRNASEWald χ2值P值HR值95%CIFLJ423510.2457.777<0.0010.420.23~0.76LINC006680.0079.938<0.0011.421.20~1.68AC136475.20.0876.237<0.0010.590.43~0.83BAIAP2-DT0.0274.853<0.0010.700.54~0.90SP2-AS10.0826.778<0.0011.651.13~2.41AC090948.30.2245.880<0.0010.500.29~0.83AL122010.10.0979.284<0.0010.470.31~0.71OTUD6B-AS10.0278.710<0.0011.631.20~2.23PIK3CD-AS20.0822.451<0.0010.670.50~0.91AC005785.10.2816.849<0.0010.420.23~0.77
表2 (续)
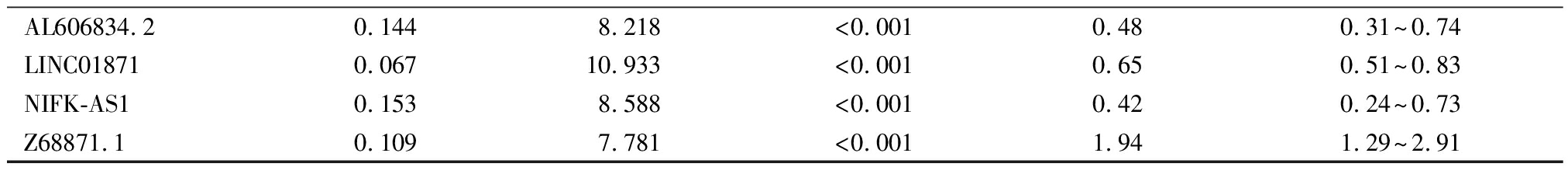
AL606834.20.1448.218<0.0010.480.31~0.74LINC018710.06710.933<0.0010.650.51~0.83NIFK-AS10.1538.588<0.0010.420.24~0.73Z68871.10.1097.781<0.0011.941.29~2.91
2.3 预后风险模型的构建和评价 使用R软件中的“survival”包对免疫相关LncRNA表达矩阵进行单因素Cox回归筛选免疫预后相关LncRNA14个,进一步纳入多因素Cox回归进一步进分析,根据最佳AIC值确定6个LncRNA建立多基因预后风险模型(图1,表3),基于所选择的LncRNA基因表达量乘以多因素Cox回归系数之和计算每例患者的风险值(risk score),根据中位风险值将乳腺癌患者分为低风险组和高风险组(图1)。采用Kaplan-Meier法对两组患者进行生存分析,并绘制生存曲线,两组乳腺癌患者预后差异有统计学意义(图2,P<0.05)。同时绘制生存预测的ROC曲线,曲线下面积AUC值为0.703(图3),显示该预后风险模型对乳腺癌患者的生存预后具有较好的预测能力。
表3 多因素Cox回归分析筛选构建预后风险模型的
免疫相关LncRNA
Table 3 Multivariate Cox regression analysis to screen
immune-related LncRNAs for constructing
prognostic risk models
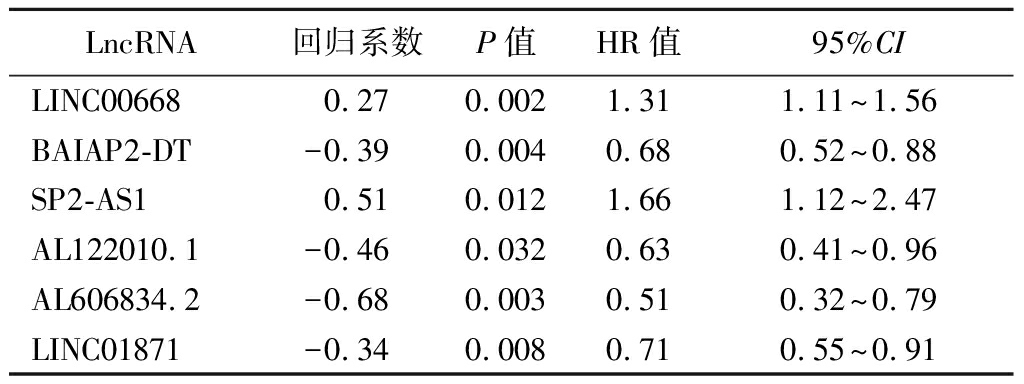
LncRNA回归系数P值HR值95%CILINC006680.270.0021.311.11~1.56BAIAP2-DT-0.390.0040.680.52~0.88SP2-AS10.510.0121.661.12~2.47AL122010.1-0.460.0320.630.41~0.96AL606834.2-0.680.0030.510.32~0.79LINC01871-0.340.0080.710.55~0.91
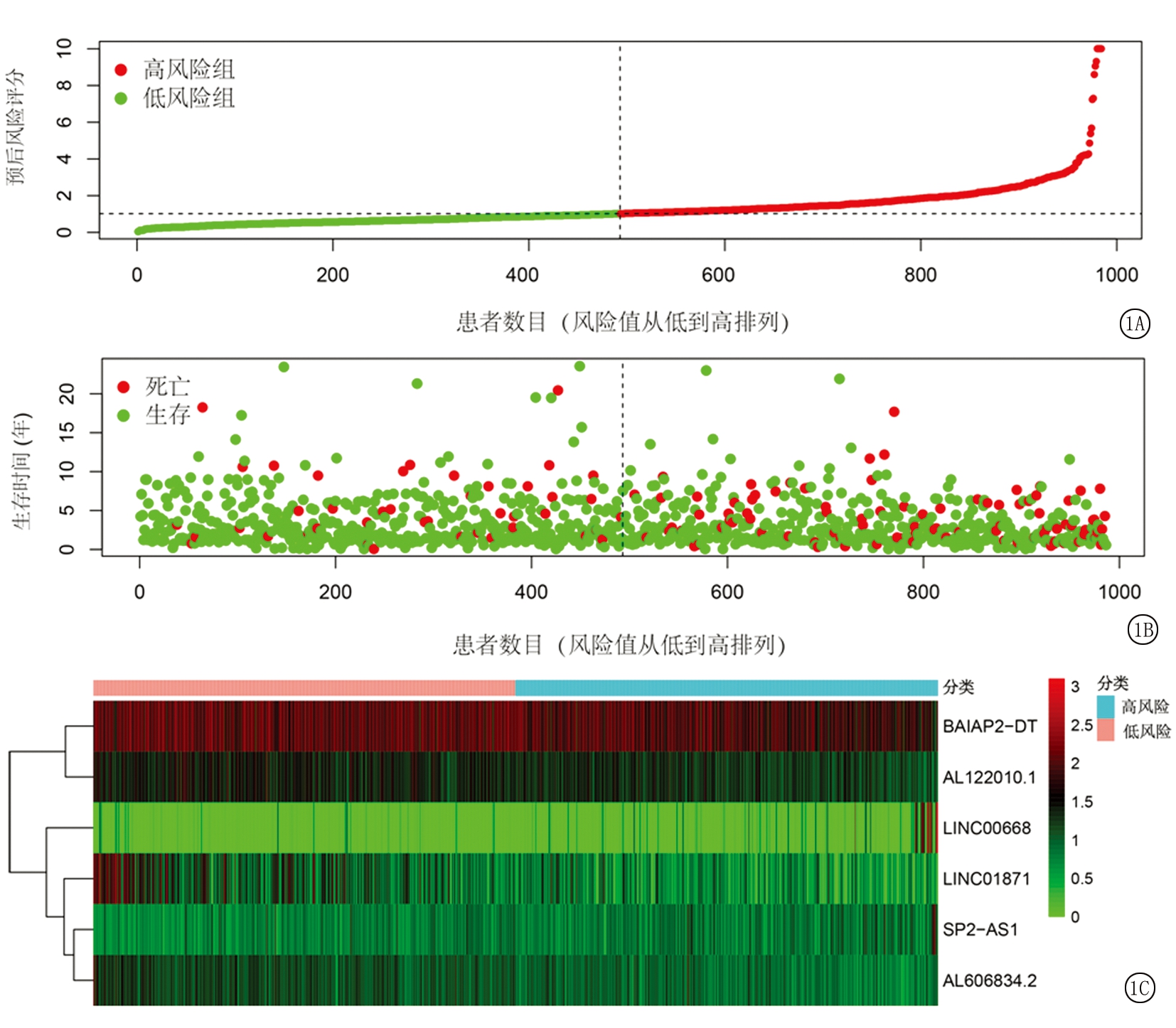
图1 预后风险模型
A.纳入的两组乳腺癌患者的风险评分;B.纳入的两组乳腺癌患者随访终点的生存状态;C.纳入预后风险模型中的6个免疫相关LncRNA基因热图
Figure Prognostic risk model
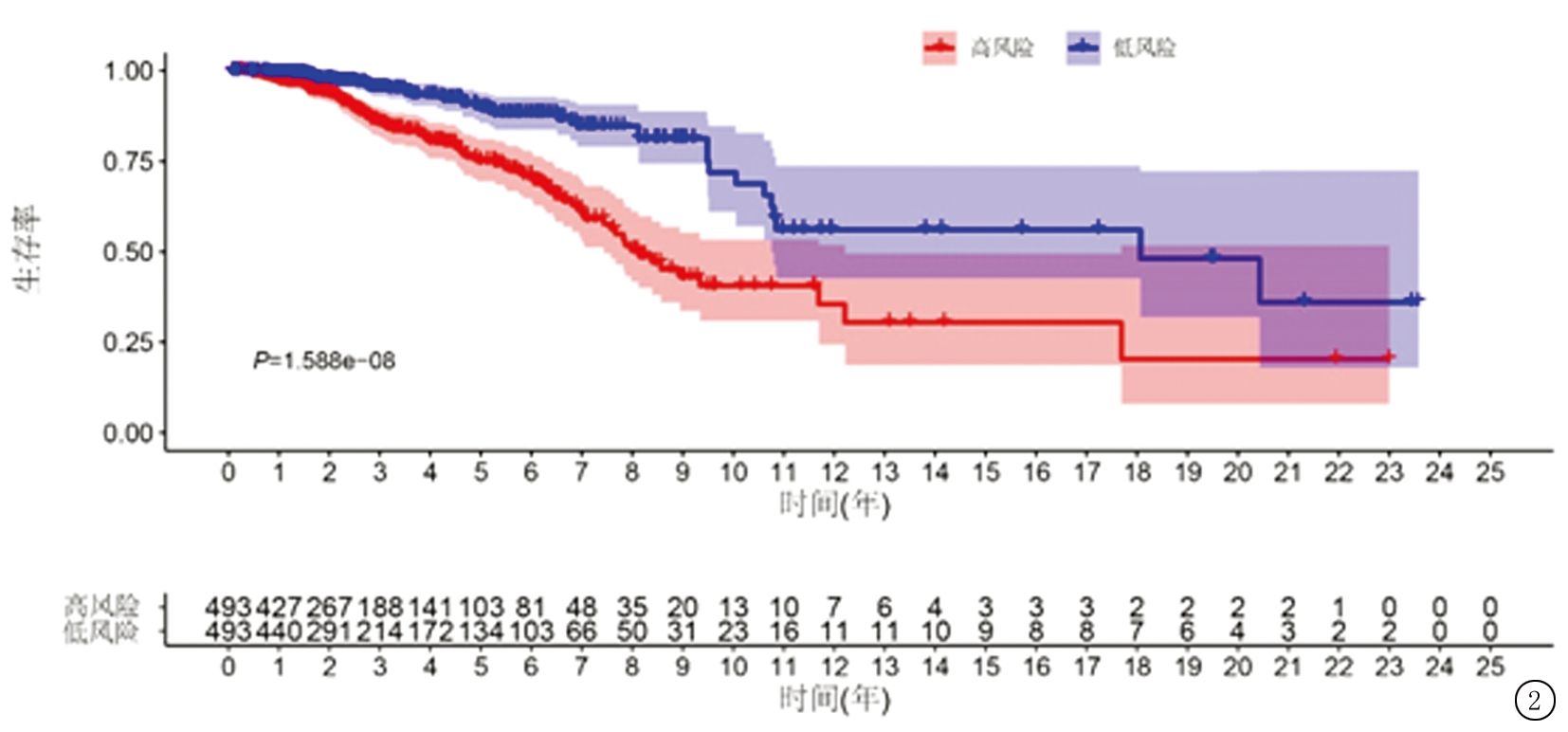
图2 两组乳腺癌患者预后风险模型Kaplan-Meier生存曲线
Figure 2 Kaplan-Meier survival curve of the prognostic risk model for the two groups of breast cancer patients
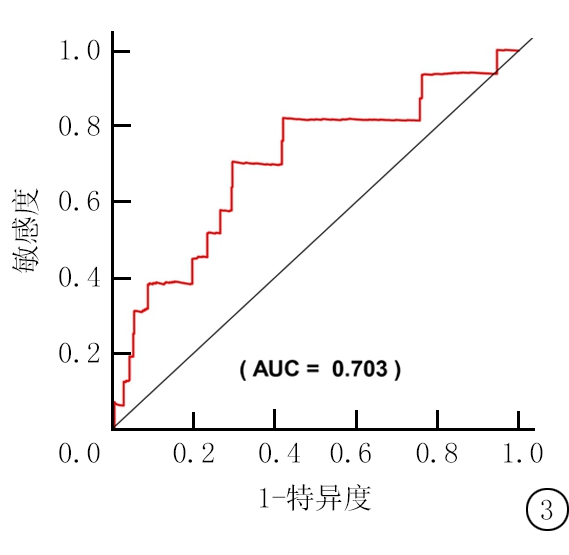
图3 乳腺癌预后风险模型的ROC曲线
Figure 3 ROC curve of breast cancer prognostic risk model
2.4 预后风险模型的临床应用 进一步验证预后风险模型在临床中的应用,结合临床病理信息及每例患者的risk score值进行单因素和多因素Cox回归分析,并绘制森林图,结果显示患者年龄和患者风险评分为乳腺癌预后的独立危险因素(P<0.05)。见图4。
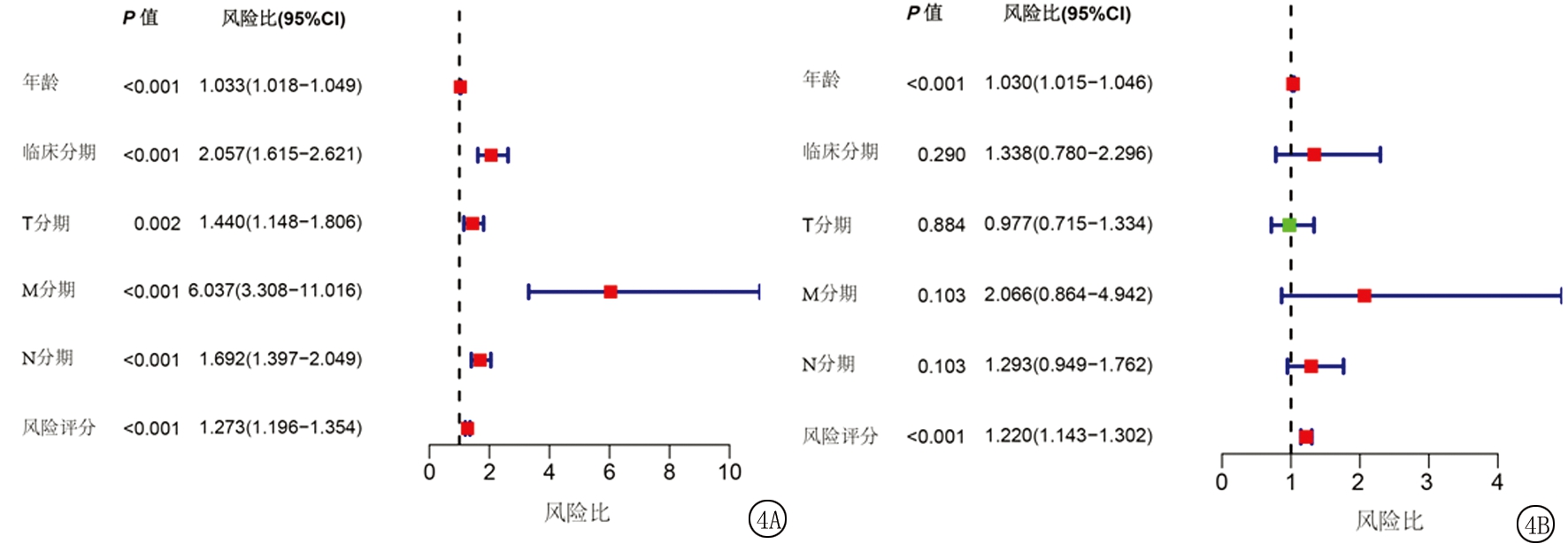
图4 乳腺癌临床病理参数及风险值Cox回归森林图
A.单因素Cox分析;B.多因素Cox分析
Figure 4 Clinicopathological parameters of breast cancer and forest plot of Cox regression analysis of risk value
3 讨 论
在我国乳腺癌的发病率已居女性恶性肿瘤的首位[2]。目前,对乳腺癌的治疗以手术、内分泌治疗、放疗、化疗和靶向治疗为主的综合治疗,使得乳腺癌的病死率较前得到明显的控制,但经过综合治疗后转移和复发仍是乳腺癌死亡的主要原因。研究显示乳腺癌是一种基因高度异质的恶性肿瘤,使得常规的临床病理评估无法精准的预测乳腺癌患者的生存预后[9]。因此寻找乳腺癌早期诊断、评估风险及预后评估的分子标志物对乳腺癌的临床治疗至关重要。随着大数据时代的到来,基因芯片技术和高通量测序获得飞速发展,精准医学也得以慢慢实现,对于肿瘤的发生发展规律也逐渐揭露。
LncRNA是指长度>200 bp并且不具备蛋白编码功能的RNA,研究发现LncRNA在肿瘤的发生、发展和肿瘤细胞的浸润转移过程中扮演着十分重要的角色,对肿瘤的早期诊断及预后评估具有重要的价值[3]。同时在多种肿瘤中研究发现免疫相关LncRNA是其生物标志物,可作为其潜在的治疗靶点,与肿瘤的生存预后显著相关[10-11]。最近在乳腺癌中也研究发现多种LncRNA 表达异常且参与其发生发展及转移的过程[5-7]。
本研究通过下载癌症基因组图谱(the cancer genome atlas,TCGA)中1 041例乳腺癌患者转录组测序数据和临床预后信息,进行全面分析,建立基因6个免疫相关LncRNA的预后风险模型。并且研究发现该模型可以较好的预测乳腺癌患者的生存预后,同时发现风险评分为乳腺癌患者预后的独立危险因素。其中6个免疫相关LncRNA中LINC00668和SP2-AS1为危险基因,BAIAP2-DT、AL122010.1、AL606834.2和LINC01871为保护基因。既往研究显示LINC00668在乳腺癌组织中高表达,通过抑制乳腺癌细胞凋亡和加速细胞周期进程来促进乳腺癌的发生发展[12]。并且有学者研究发现LINC00668通过与SND1相互作用促进乳腺癌的转移和化疗耐药性,可作为乳腺癌的潜在治疗靶点。同样也有研究发现LINC00668与乳腺癌预后显著相关[13]。Ruiz-Narv ez等[14]也研究发现BAIAP2-DT与乳腺癌单核苷酸多态性显著相关。同样Li 等[13]在乳腺癌研究中发现AL122010.1出现低表达,为乳腺癌保护基因,与其预后显著相关。在肝癌预后风险模型的研究发现LINC01871为其保护基因,作为模型构建基因与肝癌预后显著相关[15]。同样在乳腺癌的研究中也发现LINC01871为其保护基因,与其预后显著相关[13]。而SP2-AS1和AL606834.2目前尚无相关研究报道,在乳腺癌中的作用机制也尚不明确,有待进一步研究。
ez等[14]也研究发现BAIAP2-DT与乳腺癌单核苷酸多态性显著相关。同样Li 等[13]在乳腺癌研究中发现AL122010.1出现低表达,为乳腺癌保护基因,与其预后显著相关。在肝癌预后风险模型的研究发现LINC01871为其保护基因,作为模型构建基因与肝癌预后显著相关[15]。同样在乳腺癌的研究中也发现LINC01871为其保护基因,与其预后显著相关[13]。而SP2-AS1和AL606834.2目前尚无相关研究报道,在乳腺癌中的作用机制也尚不明确,有待进一步研究。
基于TCGA数据库,筛选出6个免疫相关LncRNA与乳腺癌患者预后的显著相关,并构建预后风险模型用于预测乳腺癌患者的生存预后,结合临床信息进行验证显示预后风险模型可作为乳腺癌独立危险因子,为乳腺癌独立预后生物标志物。可有效的评估乳腺癌患者的生存预后,为临床医师评估乳腺癌患者治疗疗效提供参考指标。并且预后风险模型中的6个LncRNA可能为探索乳腺癌的免疫治疗提供新的治疗靶点,同时本研究也一定的局限性,首先,这是一项回顾性研究,使用了来自TCGA数据库的数据,该数据缺乏有关治疗的信息;其次研究基于可公开获取的TCGA数据库中1 041例患者,临床数据有效从而限制了模型的预测性能;最后目前6个LncRNA在乳腺癌中分子作用机制和相关信号通路尚不清楚,仍需进一步研究。
[1] Bray F,Ferlay J,Soerjomataram I,et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin,2018,68(6):394-424.
[2] Chen W,Zheng R,Baade PD,et al. Cancer statistics in China,2015[J]. CA Cancer J Clin,2016,66(2):115-132.
[3] He RZ,Luo DX,Mo YY. Emerging roles of lncRNAs in the post-transcriptional regulation in cancer[J]. Genes Dis,2019,6(1):6-15.
[4] Quinn JJ,Chang HY. Unique features of long non-coding RNA biogenesis and function[J]. Nat Rev Genet,2016,17(1):47-62.
[5] 熊晶,王渝,王君,等.乳腺癌组织中lncRNA CCHE1水平及其临床意义[J]. 临床肿瘤学杂志,2018,23(6):519-523.
[6] 杨雪梅,李娟,王一凡,等.3-lncRNAs预后模型在HER2阳性乳腺癌预后评价中的意义[J].山东大学学报(医学版),2020,58(5):69-76.
[7] 张延涛,孙勇,张远鹏.lncRNA SNHG12在乳腺癌中的表达水平及临床意义[J].河北医药,2020,42(1):62-65.
[8] Subramanian A,Tamayo P,Mootha VK,et al. Gene set enrichment analysis:a knowledge-based approach for interpreting genome-wide expression profiles[J]. Proc Natl Acad Sci U S A,2005,102(43):15545-15550.
[9] Schettini F,Pascual T,Conte B,et al. HER2-enriched subtype and pathological complete response in HER2-positive breast cancer:a systematic review and meta-analysis[J]. Cancer Treat Rev,2020,84(3):101965-101978.
[10] Atianand MK,Caffrey DR,Fitzgerald KA. Immunobiology of long noncoding RNAs[J]. Annu Rev Immunol,2017,35(26):177-198.
[11] Xing L,Zhang X,Chen A. Prognostic 4-lncRNA-based risk model predicts survival time of patients with head and neck squamous cell carcinoma[J]. Oncol Lett,2019,18(3):3304-3316.
[12] Qiu X,Dong J,Zhao Z,et al. LncRNA LINC00668 promotes the progression of breast cancer by inhibiting apoptosis and accelerating cell cycle[J]. Onco Targets Ther,2019,11(12):5615-5625.
[13] Li X,Li Y,Yu X,et al. Identification and validation of stemness-related lncRNA prognostic signature for breast cancer[J]. J Transl Med,2020,18(1):331.
[14] Ruiz-Narv ez EA,Lunetta KL,Hong CC,et al. Genetic variation in the insulin,insulin-like growth factor,growth hormone,and leptin pathways in relation to breast cancer in African-American women:the AMBER consortium[J]. NPJ Breast Cancer,2016,2:16034.
ez EA,Lunetta KL,Hong CC,et al. Genetic variation in the insulin,insulin-like growth factor,growth hormone,and leptin pathways in relation to breast cancer in African-American women:the AMBER consortium[J]. NPJ Breast Cancer,2016,2:16034.
[15] 陈圣,赵继森,李靖华,等.肝细胞癌自噬相关长链非编码RNA预后模型的建立与分析[J].中国普通外科杂志,2020,29(7):839-848.