肝癌是常见恶性肿瘤,发病率占癌症发病率第六位[1]。肝癌的主要诱因为病毒性肝炎,但随着人们生活方式的改变,酒精相关性肝癌已占有肝癌发病的30%[2-5]。尽管采用多种治疗模式,肝癌的5年生存率仍仅为34%~50%[6-7]。早期诊断和治疗是改善预后的主要方式[8-9]。笔者回顾我院于2015—2019年收治的酒精相关性肝癌患者,发现部分患者可不经历肝硬化阶段直接进展为肝癌。寻找酒精性肝炎直接进展为肝癌的标志物可以提高此类患者的早期诊断。微小RNA(MicroRNA,miRNA)是近年来常用的肿瘤标志物,其可与目标mRNA结合,调节下游途径[10-11]。本研究旨在研究miR-151a-3p在酒精性肝炎直接进展为肝癌中的作用机制,报告如下。
1 资料与方法
1.1 一般资料 选取2015年1月1日—2019年12月31日我院收治的且经病理证实为肝细胞癌的酒精相关性肝癌患者64例的病历资料。其中男性58例,女性6例;年龄≥60岁34例,<60岁30例。根据筛选出的病历信息,于病理科随机选取5例酒精性肝炎/肝癌患者穿刺组织蜡块,并根据性别和年龄配对原则,再随机选取5例酒精性肝炎/肝硬化患者穿刺组织蜡块,比较其miR-151a-3p表达水平。
1.2 方法
1.2.1 细胞系 肝癌细胞系HepG2、Bel-7402、MHCC97、Hep3B和肝正常细胞系HL-7702从中国细胞系资源基础设施获得,由河北医科大学第四医院保留和培养,将所有细胞置于具有10%胎牛血清(Gibco,美国)的RPMI 1640培养基(Gibco,美国)中培养。
1.2.2 福尔马林固定石蜡包埋组织提取总miRNA miR特异性福尔马林固定石蜡包埋试剂盒购自比利时Qiagen分支机构。按照试剂说明进行以下操作:①将蜡块分别切成5 μm、10 μm、20 μm、30 μm、40 μm厚。根据组织的大小,将2~16片置于无RNA酶的EP试管中,向EP试管中加入1 mL二甲苯,剧烈摇动10 s,在20 ℃以14 000 r/min的速度离心2 min,丢弃上清液,根据石蜡的含量重复上述步骤1~3次。②加入1 mL的无水乙醇,摇匀并混合,以14 000 r/min的速度离心2 min,丢弃上清液。③风干,加入240 μL蛋白激酶D缓冲液和10 L蛋白酶K,摇匀并混合,根据组织溶解情况在55 ℃孵育15 min,1、12、24、36、48 h。若组织不溶解时,于24 h加入10 L蛋白酶K,继续孵育12~24 h,后在80 ℃孵育15 min。④加入500 L 红细胞裂解缓冲液,摇匀并混合,将所有液体转移至gDNA消除柱离心管中,以10 000 r/min的速度离心30 s,弃去离心管,保存上清液。⑤加入1.2 mL的无水乙醇,充分混合,将700 μL混合液转移至miR特异性试剂盒最小洗脱液离心管,以1 000 r/min的速度离心15 s,弃去上清液,重复上述过程至所有样品均离心。⑥加入500 L 藻红蛋白缓冲液,以1 000 r/min的速度离心15 s,弃去上清液,重复以上步骤,但第二次离心2 min。⑦将miR特异性试剂盒最小洗脱液离心管放入新的收集管中,以14 000 r/min的速度离心5 min,弃去上清液。⑧将miR特异性试剂盒最小洗脱液离心管置于RNA收集管中,加入30 L的无RNase离心14 000 r/min,持续1 min,以溶解总RNA(包括miRNA),使用紫外可见分光光度计测试质量和浓度。以上实验数据重复3次。
1.2.3 CCK-8实 准备90 μL肝癌细胞悬液,计数3×103个/孔,接种于96孔板中。细胞培养24 h后,向每个孔中添加10 μLLCCK-8增强溶液。于48 h和72 h时,在450 nm波长处测量吸光度(A)值,绘制曲线。以上实验数据重复3次。
1.2.4 实时定量RT-PCR RNA提取方法提取细胞总RNA(“细胞总”,指的是细胞的所有RNA),根据反转录试剂盒的说明进行操作,qPCR反应条件为50 ℃ 2 min,95 ℃ 2 min,95 ℃ 15 s,60 ℃ 15 s,40个循环和72 ℃ 1 min,数据表示为倍数差,基于2-△△Ct的计算。以上实验数据重复3次。
1.2.5 流式细胞仪测定 将肝癌细胞以1×107 个/L的密度接种于6孔板中。 培养24 h后,使用流式细胞仪检测细胞周期。以上实验数据重复3次。
1.2.6 蛋白印迹分析 提取细胞蛋白,进行十二烷基硫酸钠电泳,转移到聚偏二氟乙烯膜上,孵育第一抗体过夜,并孵育第二抗体4 h,以增强化学发光超敏发光液发展。以上实验数据重复3次。
1.2.7 在肝癌细胞系中敲除miR-151a-3p 将肝癌细胞接种在6孔细胞培养皿中,并用慢病毒(si-miR-151a-3p)转导24 h。于48 h使用0.8 mg/L嘌呤霉素进行选择,直至平行实验的所有亲代细胞死亡,剩余活细胞是具有敲低的miR-151a-3p的稳定的HCC细胞系。本研究中使用的敲除miR-151a-3p的序列是5′-CUAGACUGAAGCUCCUUGAG-G-3′(GeneChem,中国上海)。被miR-151a-3p击倒的稳定细胞系即为si-miR-151a-3p。以上实验数据重复3次。
1.2.8 荧光素酶报告实验 预测ATM的3′-非翻译区序列与miR-151a-3p相互作用,或合成预测的靶位点内的突变序列并将其插入pGL3对照荧光素酶报道载体(Promega,美国威斯康星州)。后产生pGL3-ATM-Wt和pGL3-ATM-Mut载体。将miR-151a-3p模拟物、pGL3、pGL3-ATM-Mut、pGL3-ATM-Wt质粒共转染肝癌细胞48 h。随后裂解细胞,通过荧光素酶检测试剂盒(Promega)分别测定荧光素酶活性。以上实验数据重复3次。
1.3 统计学方法 应用SPSS 19.0统计软件分析数据。计量资料比较采用t检验、单因素方差分析、SNK-q检验,计数资料比较采用χ2检验,采用Fisher精确概率法确定miRNA与患者信息变量之间的关系。P<0.05为差异有统计学意义。
2 结 果
2.1 酒精性肝癌患者的一般资料 所有酒精相关性肝癌患者均由病理证实为肝细胞癌,且除外感染病毒性肝炎。其中,酒精性肝炎经酒精性肝硬化最终进展为肝癌44例;酒精性肝炎未经历肝硬化阶段直接进展为肝癌20例。酒精性肝炎/肝癌患者年龄较酒精性肝炎/肝硬化/肝癌患者年轻,差异有统计学意义(P<0.05)。见表1。
表1 不同进展过程酒精相关性肝癌患者临床比较
Table 1 Clinical comparison of patients with alcohol-related
liver cancer in different progression stages (例数)
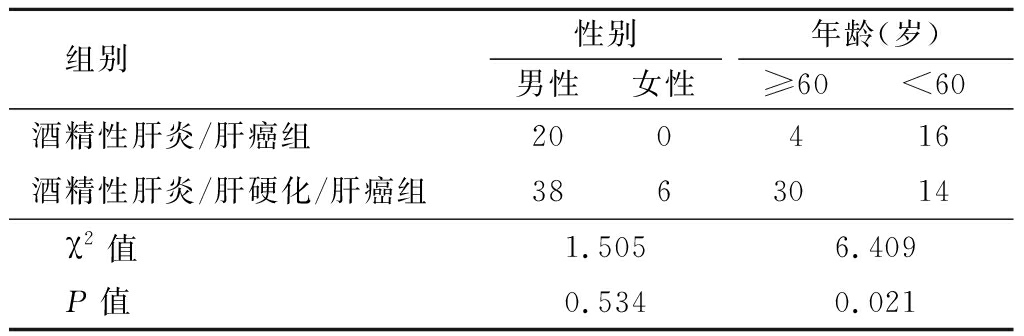
组别性别男性女性年龄(岁)≥60<60酒精性肝炎/肝癌组200416酒精性肝炎/肝硬化/肝癌组3863014 χ2值1.5056.409 P值0.5340.021
2.2 两组患者癌组织中miR-151a-3p表达水平比较 酒精性肝炎/肝癌患者癌组织中miR-151a-3p表达水平2.377±0.441显著高于酒精性肝炎/肝硬化患者肝组织1.146±0.299,差异有统计学意义(t=6.989,P=0.002)。
2.3 肝癌细胞系中miR-151a-3p表达水平升高 评估miR-151a-3p在4种肝癌细胞系和1种正常肝细胞系中的表达,结果表明miR-151a-3p 在4种常见肝癌细胞系中的表达显著高于正常肝细胞系。而在肝癌细胞系中,HepG2中的表达最高。见表2。
表2 miR-151a-3p在不同肝癌细胞系和
正常肝细胞系中的表达水平
Table 2 The expression level of miR-151a-3p in different
liver cancer cell lines and normal liver cell lines ![]()
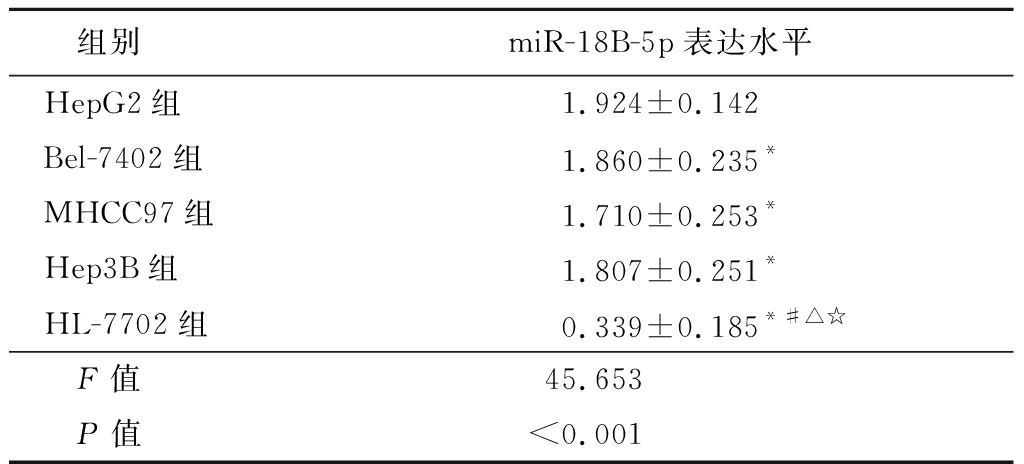
组别miR-18B-5p表达水平HepG2组1.924±0.142Bel-7402组1.860±0.235*MHCC97组1.710±0.253*Hep3B组1.807±0.251*HL-7702组0.339±0.185*#△☆ F值45.653 P值<0.001
*P值<0.05与HepG2组比较 #P值<0.05与Bel-7402组比较 △P值<0.05与MHCC97组比较 ☆P值<0.05与Hep3B组比较(SNK-q检验)
2.4 下调miR-151a-3p的影响 下调HepG2和Bel-7402中miR-151a-3p的表达水平,Si-miR-151a-3p(HepG2)细胞系和Si-miR-151a-3p(Bel-7402)细胞系中G0/G1期的细胞百分比明显增加,而 S期和G2/M期的细胞明显减少。见表3,4。
表3 HepG2细胞和Bel-7402细胞与其下调
miR-151a-3p表达后miR-151a-3p表达水平比较
Table 3 Comparison of miR-151a-3p expression levels in
HepG2 cells and Bel-7402 cells and those after
down-regulation of miR-151a-3p expression cells![]()
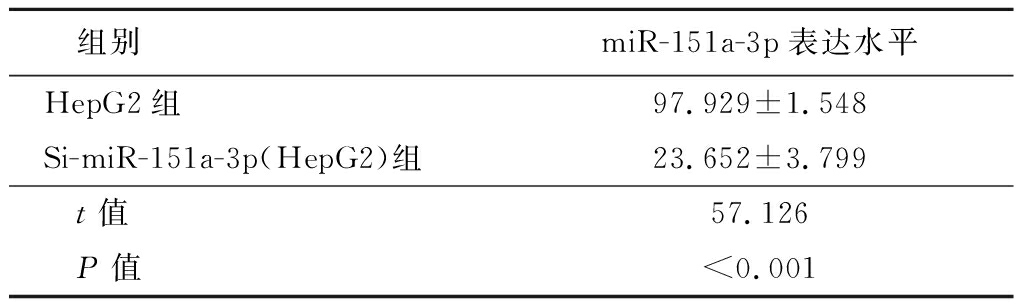
组别miR-151a-3p表达水平HepG2组97.929±1.548Si-miR-151a-3p(HepG2)组23.652±3.799 t值57.126 P值<0.001
表4 Bel-7402细胞与其下调miR-151a-3p表达后
miR-151a-3p表达水平比较
Table 4 Comparison of miR-151a-3p expression levels in
Bel-7402 cells and those after down-regulation of
miR-151a-3p expression cells![]()
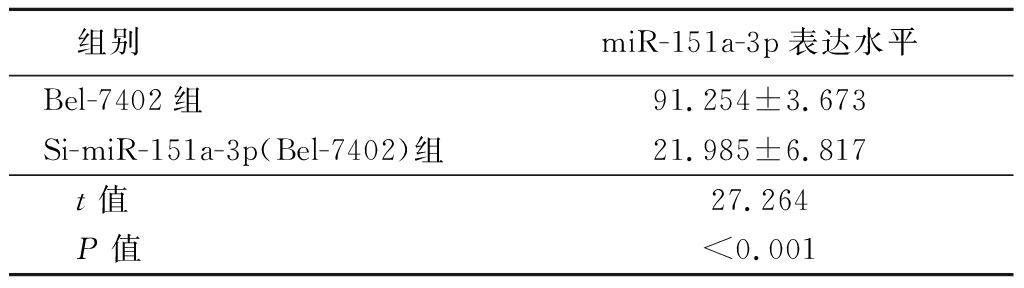
组别miR-151a-3p表达水平Bel-7402组91.254±3.673Si-miR-151a-3p(Bel-7402)组21.985±6.817 t值27.264 P值<0.001
2.5 下调miR-151a-3p抑制细胞周期的机制 与HepG2比较,Si-miR-151a-3p(HepG2)细胞中ATM的mRNA和蛋白表达水平增加,P53的mRNA表达水平增加,P-P53蛋白的表达水平增加,而Cyclin D1的mRNA和蛋白的表达水平下降,差异有统计学意义(P<0.05)。同样,与Bel-7402相比,Si-miR-151a-3p(Bel-7402)细胞中ATM的mRNA表达水平显著增加,P53的mRNA表达水平显着增加,而Cyclin D1的mRNA的表达水平显着下降。见表5~8。
表5 HepG2细胞系ATM 、P53、Cyclin D1 mRNA表达水平
Table 5 The mRNA expression levels of ATM, P53, and Cyclin D1 in HepG2 cell lines![]()
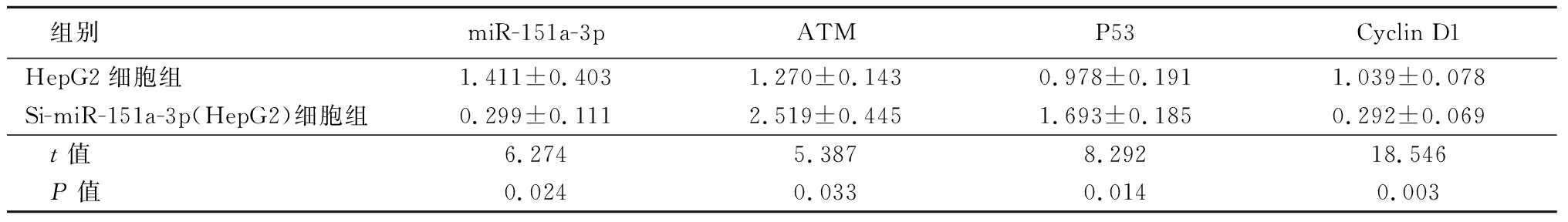
组别miR-151a-3pATMP53Cyclin D1HepG2细胞组1.411±0.4031.270±0.1430.978±0.1911.039±0.078Si-miR-151a-3p(HepG2)细胞组0.299±0.1112.519±0.4451.693±0.1850.292±0.069 t值6.2745.3878.29218.546 P值0.0240.0330.0140.003
表6 Bel-7402细胞系ATM 、P53、Cyclin D1 mRNA表达水平
Table 6 The mRNA expression levels of ATM, P53, and Cyclin D1 in Bel-7402 cell lines![]()
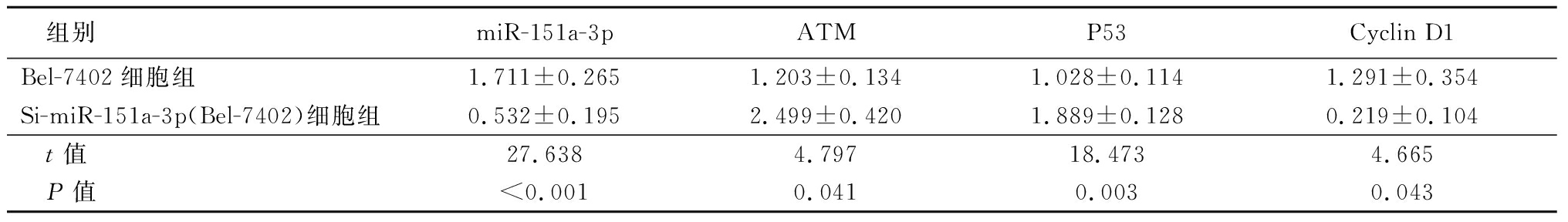
组别miR-151a-3pATMP53Cyclin D1Bel-7402细胞组1.711±0.2651.203±0.1341.028±0.1141.291±0.354Si-miR-151a-3p(Bel-7402)细胞组0.532±0.1952.499±0.4201.889±0.1280.219±0.104 t值27.6384.79718.4734.665 P值<0.0010.0410.0030.043
表7 HepG2细胞系ATM 、P53、Cyclin D1 蛋白表达水平
Table 7 The protein expression levels of ATM, P53, and Cyclin D1 in HepG2 cell lines![]()

组别ATMP-P53Cyclin D1HepG2细胞组0.072±0.0260.074±0.0090.747±0.130Si-miR-151a-3p(HepG2)细胞组0.756±0.0790.449±0.1080.224±0.053 t值11.7136.54311.841 P值0.0070.0230.007
表8 Bel-7402细胞系ATM 、P53、Cyclin D1 蛋白表达水平
Table 8 The protein expression levels of ATM, P53, and Cyclin D1 in Bel-7402 cell lines![]()

组别ATMP-P53Cyclin D1Bel-7402细胞组0.058±0.0230.064±0.0090.647±0.133Si-miR-151a-3p(Bel-7402)细胞组0.823±0.1230.716±0.1310.111±0.037 t值11.5228.0959.568 P值0.0070.0150.011
2.6 miR-151a-3p调节细胞周期的机制 荧光素酶报告实验结果显示,在HepG2细胞系和Bel-7402细胞系中,相应的ATM-Mut构建体不再被miR-151a-3p抑制,表明miR-151a-3p是直接靶向ATM-WT的miRNA。见图2、表9,10。
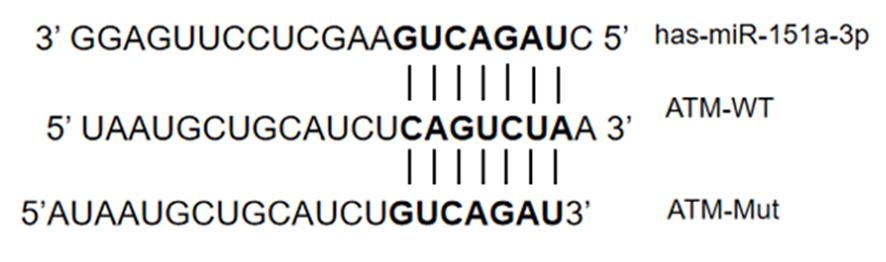
图1 ATM RNA的假定的miR-151a-3p结合序列
Figure 1 Putative miR-151a-3p binding sequence for ATM RNA
表9 qRT-PCR检测HepG2细胞系和Si-miR-
151a-3p(HepG2)中ATM基因表达水平
Table 9 qRT-PCR detection of ATM gene expression
level in HepG2 cell lines and Si-miR-151a-3p
(HepG2)cell lines![]()
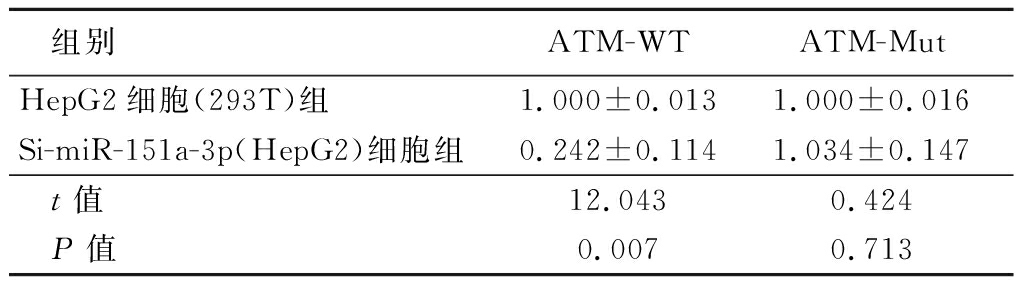
组别ATM-WTATM-MutHepG2细胞(293T)组1.000±0.0131.000±0.016Si-miR-151a-3p(HepG2)细胞组0.242±0.1141.034±0.147 t值12.0430.424 P值0.0070.713
表10 qRT-PCR检测Bel-7402细胞系和
Si-miR-151a-3p(Bel-7402)中ATM基因表达水平
Table 10 qRT-PCR detection of ATM gene expression level
in Bel-7402 cell lines and Si-miR-151a-3p
(Bel-7402)cell lines![]()
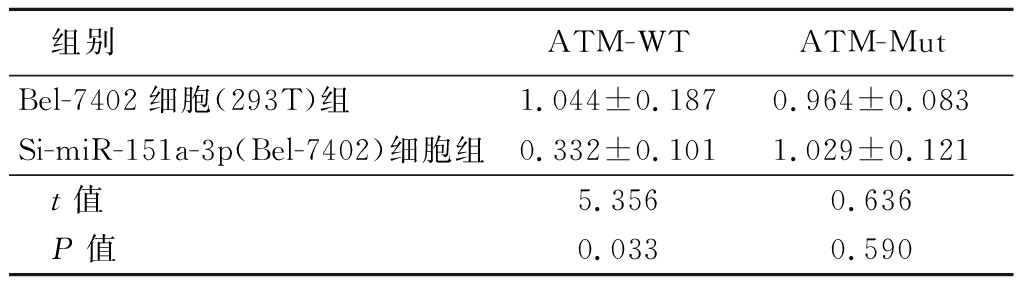
组别ATM-WTATM-MutBel-7402细胞(293T)组1.044±0.1870.964±0.083Si-miR-151a-3p(Bel-7402)细胞组0.332±0.1011.029±0.121 t值5.3560.636 P值0.0330.590
3 讨 论
大量长期饮酒对人体的DNA、蛋白质造成永久性损伤,增加促炎细胞因子释放,减少抗肿瘤细胞的CD8+T细胞招募,从而促进免疫反应的抑制,导致肝癌的发生[12]。研究酒精性肝炎进展至肝癌的过程,有助于实现肝癌的早期诊断和治疗。本研究于我院病案室筛选住院治疗且经病理证实为肝细胞癌的酒精相关性肝癌患者64例,其中44例为酒精性肝炎经酒精性肝硬化,最终进展为肝癌;20例为酒精性肝炎,未经历肝硬化阶段,直接进展为肝癌。对比临床病历特征,酒精性肝炎/肝癌患者年龄较酒精性肝炎/肝硬化/肝癌患者年轻。这说明其可能经历了不同的进展过程。
本研究对比5例酒精性肝炎/肝癌患者组织和5例酒精性肝炎/肝硬化患者组织中10种相关miRNA的表达,结果显示,酒精性肝炎/肝癌患者组织中miR-151a-3p的表达水平显著高于酒精性肝炎/肝硬化患者。因此,笔者认为miR-151a-3p可能在酒精性肝炎直接进展为肝癌的发病过程中起到重要作用。
为了进一步探讨miR-151a-3p在酒精相关性肝癌中的作用,本研究进行了体外实验。首先,验证miR-151a-3p在肝癌组织中的表达水平。在4种常见的HCC细胞系和1种肝正常细胞系中检测miR-151a-3p的表达水平,结果显示,miR-151a-3p在4种肝癌细胞系中的表达均显著高于肝正常细胞系,且在HepG2中的表达水平最高。本研究在2种肝癌细胞系中验证miR-151a-3p的作用机制。在HepG2和Bel-7402中通过慢病毒下调miR-151a-3p表达水平,结果显示,Si-miR-151a-3p(HepG2和Bel-7402)细胞系G0/G1期的细胞百分比显着增加,而 S期和G2/M期的细胞明显减少。实验结果表明miR-151a-3p可以通过促进肝癌细胞进入细胞周期,从而促进肝癌的发生与发展。但其具体机制尚需进一步探索。
Liu等[13]研究显示,miR-151a-3p可以通过抑制p53来促进鼻咽癌的增殖,迁移和侵袭。因此,本研究使用qRT-PCR和蛋白印迹分析检测与细胞周期相关蛋白的mRNA和蛋白表达水平,结果表明,在Si-miR-151a-3p(HepG2和Bel-7402)细胞系中,ATM的mRNA和蛋白表达水平显著增加,P53的mRNA表达水平显着增加,P-P53蛋白的表达水平显著增加,而Cyclin D1的mRNA和蛋白的表达水平显着下降。共济失调毛细血管扩张突变是一种丝氨酸/苏氨酸激酶,通常被认为是一种肿瘤抑制因子[14-15]。ATM被激活以磷酸化不同细胞周期中的不同靶蛋白。P53是ATM基因的下游基因,是细胞信号通路的重要组成部分,主要在抑制细胞癌中发挥作用[16]。 P53抗肿瘤作用最重要的方法是阻滞细胞周期并诱导细胞凋亡[17-19]。此外,本研究构建了荧光素酶报告分子,显示相应的ATM-Mut构建体不再受到miR-151a-3p的抑制,说明miR-151a-3p可以通过直接靶向ATM从而抑制其表达。本研究显示miR-151a-3p在肝癌细胞中过表达,通过直接靶向ATM抑制P53,从而促进cyclin D1的表达,导致肝癌的进展。
综上所述,部分酒精性肝炎患者可以不经过肝硬化阶段而直接进展为肝癌。miR-151a-3p在其中扮演重要角色,通过直接靶向ATM抑制P53,从而促进Cyclin D1的表达,导致肝癌发生、发展。
[1] Bray F,Ferlay J,Soerjomataram I,et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin,2018,68(6):394-424.
[2] Ke LX,Shen R,Fan WZ,et al. The role of associating liver partition and portal vein ligation for staged hepatectomy in unresectable hepatitis B virus-related hepatocellular carcinoma[J]. Ann Transl Med,2020,8(21):1402.
[3] van Mierlo KM,Schaap FG,Dejong CH,et al. Liver resection for cancer:New developments in prediction,prevention and management of postresectional liver failure[J]. J Hepatol,2016,65:1217-1231.
[4] El-Serag HB. Epidemiology of viral hepatitis and hepatocellular carcinoma[J]. Gastroenterology,2012,142(6):1264-1273.
[5] Laura P,Helena CP,Francesco N,et al. Burden of liver disease in Europe:Epidemiology and analysis of risk factors to identify prevention policies[J]. J Hepatol,2018,69(3):718-735.
[6] Liu PH,Hsu CY,Hsia CY,et al. Surgical resection versus radiofrequency ablation for single hepatocellular carcinoma ≤ 2 cm in a propensity score model[J]. Ann Surg,2016,263(3):538-545.
[7] Ponziani FR,Bhoori S,Castelli C,et al. Hepatocellular carcinoma is associated with gut microbiota profile and inflammation in nonalcoholic fatty liver disease[J]. Hepatology,2019,69(1):107-120.
[8] Jorge AM,Laura MK,Claude BS,et al. Diagnosis,staging,and management of hepatocellular carcinoma:2018 practice guidance by the American Association for the study of liver diseases[J]. Hepatology,2018,68(2):723-750.
[9] Walid SA,Justin S,Ju DY,et al. Current status of hepatocellular carcinoma detection:screening strategies and novel biomarkers[J]. Ther Adv Med Oncol,2019,11:1758835919869120.
[10] Vescarelli E,Gerini G,Megiorni F,et al. MiR-200c sensitizes Olaparib-resistant ovarian cancer cells by targeting Neuropilin 1[J]. J Exp Clin Cancer Res,2020,39(1):3.
[11] Fu JX,Zhao JH,Zhang HM,et al. MicroRNA-451a prevents cutaneous squamous cell carcinoma progression via the 3-phosphoinositide-dependent protein kinase-1-mediated PI3K/AKT signaling pathway[J]. Exp Ther Med,2021,21(2):116.
[12] 丁梦梦.高沿航.IL-17信号通路在酒精相关性肝癌发生、发展中的作用[J].肝脏,2020,25(6):562-564.
[13] Liu HB,Cheng Y,Xu YP,et al. The inhibition of tumor protein p53 by microRNA-151a-3p induced cell proliferation,migration and invasion in nasopharyngeal carcinoma[J]. Biosci Rep,2019,39(10):BSR20191357.
[14] Zhao MH,Wang YW,Zhao YC,et al. Caspase-3 knockout attenuates radiation-induced tumor repopulation via impairing the ATM/p53/Cox-2/PGE 2 pathway in non-small cell lung cancer[J]. Aging(Albany NY),2020,12(21):21758-21776.
[15] Wang P,Wang C,Liu CY. Antitumor effects of dioscin in A431 cells via adjusting ATM/p53-mediated cell apoptosis,DNA damage and migration[J]. Oncol Lett,2021,21(1):59.
[16] Khanna KK,Lavin MF,Jackson SP,et al. ATM,a central controller of cellular responses to DNA damage[J]. Cell Death Differ,2001,8(11):1052-1065.
[17] Bieging-Rolett KT,Attardi LD. Zmat3 splices together p53-dependent tumor suppression[J]. Mol Cell Oncol,2021,8(3):1898523.
[18] Böhlig L,Rother K. One function-multiple mechanisms:the manifold activities of p53 as a transcriptional repressor[J]. J Biomed Biotechnol,2011,2011:464916.
[19] Bunz F,Dutriaux A,Lengauer C,et al. Requirement for p53 and p21 to sustain G2 arrest after DNA damage[J]. Science,1998,282(5393):1497-1501.