Peutz-Jeghers综合征(Peutz-Jeghers syndrome,PJS)是一种罕见的常染色体显性遗传疾病,发病率为1/50 000~1/200 000 [1]。因该病发病率极低,有关PJS的报道多为病例报告、病例系列和病例回顾,而包含大量病例的队列研究则很少。该病以胃肠道多发性息肉、皮肤黏膜色素沉着斑和肿瘤易感性为主要特征[2]。目前PJS诊断要素包括:胃肠道错构性息肉、皮肤黏膜色素沉着斑和疾病家族史[3-4]。因PJS患者具有明显的家族聚集性,且恶性肿瘤风险比一般人群高9~18倍,尤其大肠癌的肿瘤终生风险可达39%[5],故该病被纳入遗传性结直肠癌综合征[4]之中,如Lynch综合征、家族性腺瘤性息肉病等。随着近年来研究的深入,多位学者研究发现不同突变位点及类型,可能影响PJS的临床表现,尤其恶性肿瘤发生率[6-7]。在PJS患者中连续发现STK11突变,并确定丝氨酸-苏氨酸激酶11(STK11 / LKB1,OMIM 602216)为PJS的主要致病基因。因检测数量和采用的检测方式不同,导致致突变检出率差异较大。目前检测方法包括Sanger测序、二代测序(next-generation sequencing,NGS)和多重连锁依赖性探针扩增分析(multiplex ligation-dependent probe amplification,MLPA)等。其中,Sanger测序联合MLPA或NGS联合MLPA是目前为止最好的组合检测策略,检出率为60%~100%[2]。这些被检测到的突变会被记录在孟德尔遗传在线(online mendelian inheritance in man,OMIM)、人类基因突变数据库(human gene mutation database,HGMD)、ClinVar等数据库中,临床医生可通过检索并进行基因型-表型关联、遗传咨询等相关工作。但是,随着测序技术进步和突变命名规则确立等多种原因,数据库所记录的突变中存在的问题逐渐被认识。其中部分问题可能会影响上述临床工作,如对PJS患者致病突变的鉴定和由此展开的遗传咨询,还会导致新发突变的错误报道,进而引起后续的报道错误。为了探究这些问题的发生原因,本研究对中国PJS患者中报道的STK11突变进行了全面检索及分析。希望通过揭示该现象发生的原因,为新发现突变的标准表达提供参考。
1 资料与方法
1.1 一般资料 检索Medline、PubMed、Embase、万方数据库、中国期刊全文数据库(CNKI)、中国生物医学文献数据库(CBM & SinoMed)。检索年限:从创刊至2020年12月31日。中文主题词:“中国或中国人、Peutz-Jeghers综合征或PJS、STK11或丝氨酸/苏氨酸11或LKB1或肝激酶B1)”,英文主题词:“China/Chinese、Peutz-Jeghers syndrome/PJS、STK11或Serine/threonine kinase 11或LKB1或Liver kinase B1”。此外对部分文献进行人工检索和核对交叉引用。
1.2 纳入标准和排除标准 纳入标准:①包括队列研究、病例对照研究和病例报告/病例系列,其中所报道中国的PJS患者均已明确诊断,并完成了对STK11基因的突变检测;②突变检测方法包括Sanger序列、全外显子测序和MLPA等;③检测对象包括PJS患者本人及其亲属,以及因发现胃肠道息肉而疑诊为PJS的患者等。排除标准:①对PJS患者仅围绕临床诊断、临床特征、治疗、预后等临床过程进行讨论的研究;②未经同行评审的学位论文;③采用错误STK11基因序列进行突变命名者。
1.3 定性分析和数据 提取对纳入的研究进行多方面的质量评估,包括患者来源、实验方法和报告形式,评估由两名研究人员独立进行。如果研究或案例的质量被认为不合格,则将其从以下分析中排除。如有任何分歧,应由调查员进行判断。从最终纳入的研究中提取以下有关变量:第一作者的姓氏,进行研究的时间和地点;突变检出率;受检的家系与检出家系数量;突变的具体信息(外显子、编码区的位置、氨基酸改变);突变的性质(未报道、已报道);存在问题突变的细节。
1.4 结果分析 采用描述性分析。结果包括中国PJS患者所携带的STK11突变,报告时间和地点,突变数及其性质以及报告错误和原因。为了对其进行判断,检索相关数据库和文献以确认所报道突变是否为新突变。
2 结 果
2.1 文献检索结果及基本特征 按照上述检索范围、纳入和排除标准进行检索和筛选,共纳入55篇文献,经统计显示,包含649个家系(一篇文献因不能准确识别参检和检出家系数据未能参与家系统计),共报道301个突变,包括一些重复报道的突变。经过系统分析,最终确定181种突变,其中中国学者首次报道108种突变(图1)。包含55条文章的具体信息见表1。迄今为止,HGMD中记录了412种不同的STK11突变,这是目前为止可检索最全面的PJS致病突变数据库,中国PJS患者携带的突变占比26.2%。
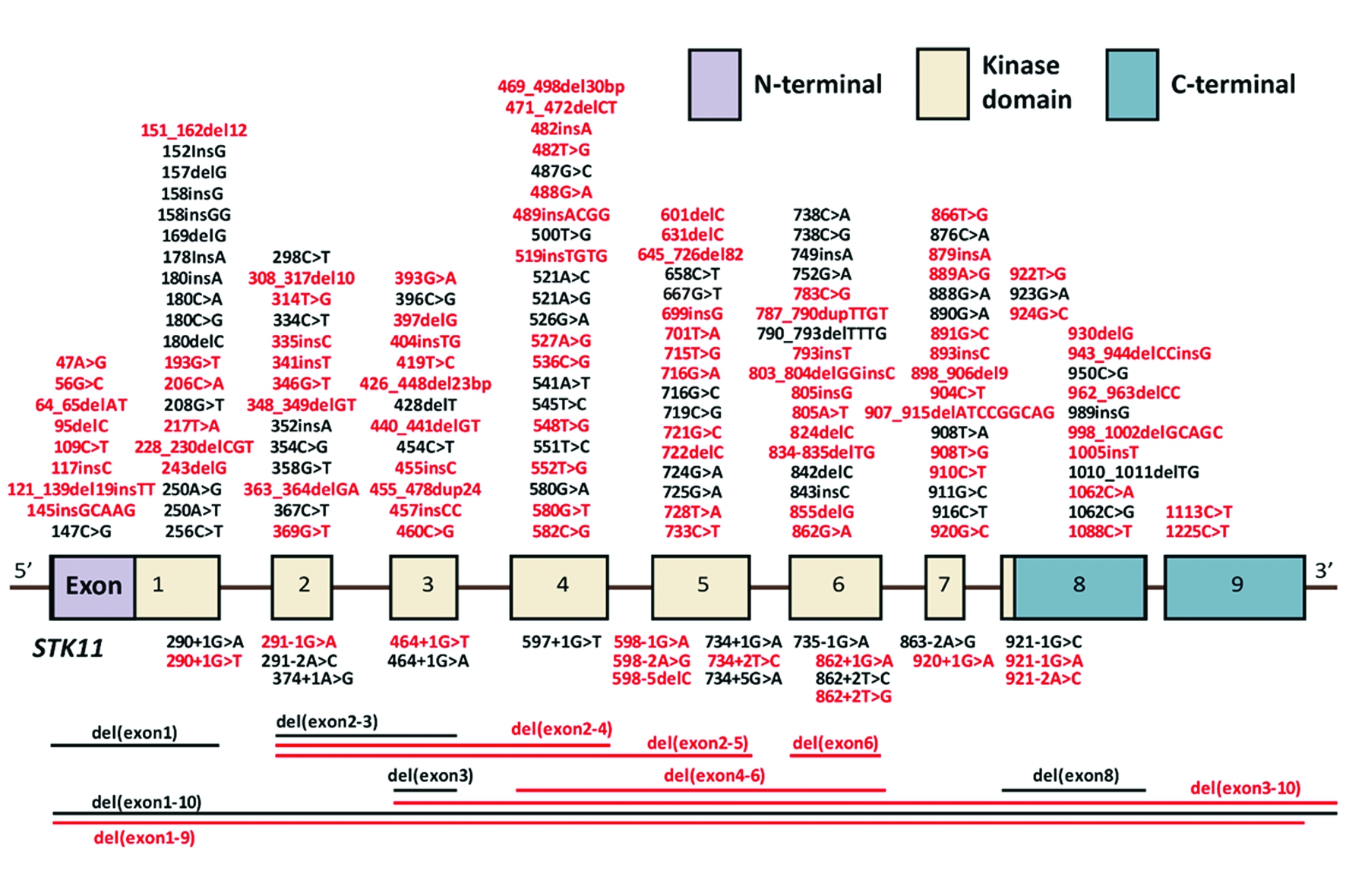
图1 文献报道中国PJS患者STK11突变基因分布图
Figure 1 Distribution of STK11 mutations in Chinese PJS patients in literature review
表1 文献报道中国PJS患者STK11突变汇总情况
Table 1 Information of articles reporting STK11 mutation in Chinese PJS patients
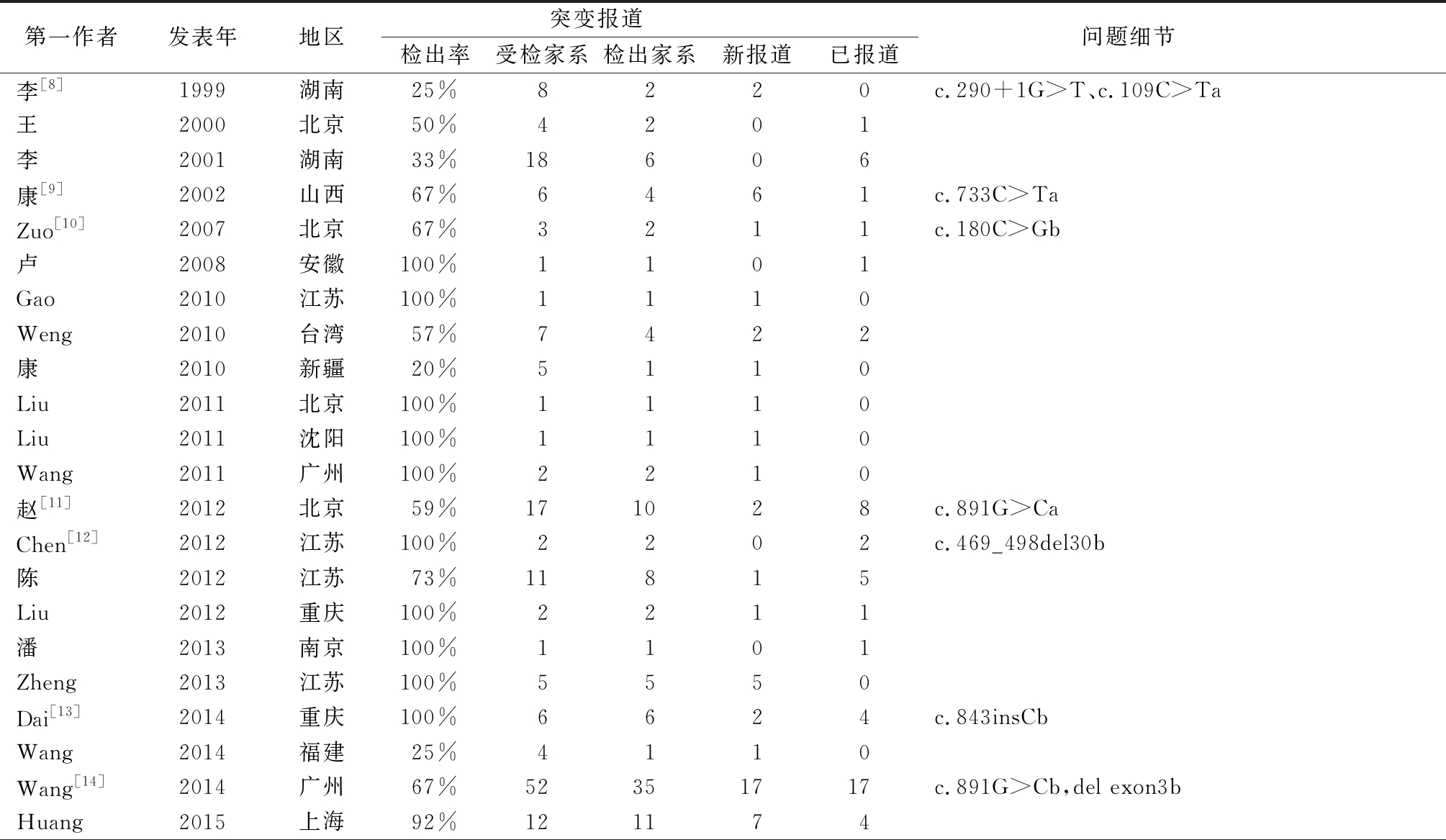
第一作者发表年地区突变报道检出率受检家系检出家系新报道已报道问题细节李[8]1999湖南25%8220c.290+1G>T、c.109C>Ta王2000北京50%4201李2001湖南33%18606康[9]2002山西67%6461c.733C>TaZuo[10]2007北京67%3211c.180C>Gb卢2008安徽100%1101Gao2010江苏100%1110Weng2010台湾57%7422康2010新疆20%5110Liu2011北京100%1110Liu2011沈阳100%1110Wang2011广州100%2210赵[11]2012北京59%171028c.891G>CaChen[12]2012江苏100%2202c.469_498del30b陈2012江苏73%11815Liu2012重庆100%2211潘2013南京100%1101Zheng2013江苏100%5550Dai[13]2014重庆100%6624c.843insCbWang2014福建25%4110Wang[14]2014广州67%52351717c.891G>Cb,del exon3bHuang2015上海92%121174
表1 (续)
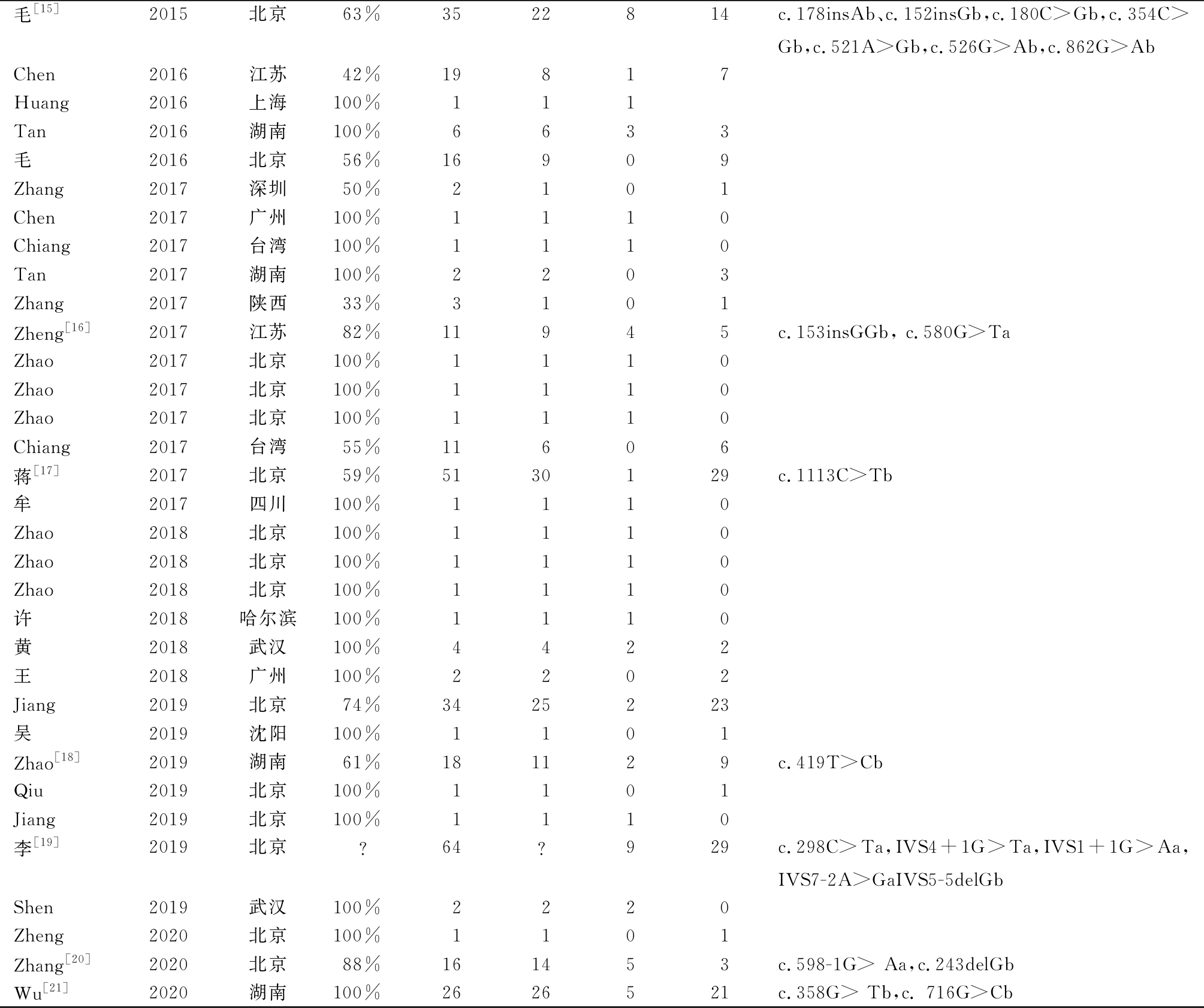
a:新发突变报道错误;b:重复报道;?:该文章未提及具体先证者数量,无法测算检出率
毛[15]2015北京63%3522814c.178insAb、c.152insGb,c.180C>Gb,c.354C>Gb,c.521A>Gb,c.526G>Ab,c.862G>AbChen2016江苏42%19817Huang2016上海100%111Tan2016湖南100%6633毛2016北京56%16909Zhang2017深圳50%2101Chen2017广州100%1110Chiang2017台湾100%1110Tan2017湖南100%2203Zhang2017陕西33%3101Zheng[16]2017江苏82%11945c.153insGGb, c.580G>TaZhao2017北京100%1110Zhao2017北京100%1110Zhao2017北京100%1110Chiang2017台湾55%11606蒋[17]2017北京59%5130129c.1113C>Tb牟2017四川100%1110Zhao2018北京100%1110Zhao2018北京100%1110Zhao2018北京100%1110许2018哈尔滨100%1110黄2018武汉100%4422王2018广州100%2202Jiang2019北京74%3425223吴2019沈阳100%1101Zhao[18]2019湖南61%181129c.419T>CbQiu2019北京100%1101Jiang2019北京100%1110李[19]2019北京?64?929c.298C>Ta,IVS4+1G>Ta,IVS1+1G>Aa,IVS7-2A>GaIVS5-5delGbShen2019武汉100%2220Zheng2020北京100%1101Zhang[20]2020北京88%161453c.598-1G> Aa,c.243delGbWu[21]2020湖南100%2626521c.358G> Tb,c. 716G>Cb
2.2 突变报道错误统计 共发现27种错误报道(其中22种重复报道和5种新突变命名错误),占本文报道的所有病例的25%。导致命名错误原因包括:数据库收录不全,突变识别错误。重复报道原因包括:文献综述不足,因错误信息导致的后续错误报道,短期内重复报道,以及因重复序列而命名错误。见表2。因重复序列而命名错位,是因为发生插入突变的位点位于重复序列之间,导致命名错误。为了证实上述情况,对其中带有c.178insA,c.152insG突变PJS患者的STK11基因相应区域进行反向测序,并检测到这两个突变位点(图2)。分别自178和152位开始前方的碱基序列出现双峰,提示碱基插入位置在该两个位点之前。并最终证实了正确命名方式为c.180insA和c.158insG。
表2 STK11突变报道错误原因汇总
Table 2 Summary of causes of error reporting on STK11 mutations

突变错报原因突变信息命名错误原因 数据库收录不全c.290+1G>T,c.109C>T,c.733C>T 突变识别错误c.891G>C,c.580G>T重复报道原因 文献综述不足c.180C>G,c.180C>G,c.354C>G,c.521A>G, c.526G>A,c. 862G>A,c.298C>T,IVS4+1G>T,IVS1+1 G>A,IVS7-2 A>G,c.598-1G> A,c.358G> T 因错误信息导致的后续错误报道891G>C 短期内重复报道c.469_498del30nt,del exon3,c.862G>A,c.419T>C,c.243delG 因重复序列引起的命名错误c.843insC,c.178insA,c.152insG,c.153insGG
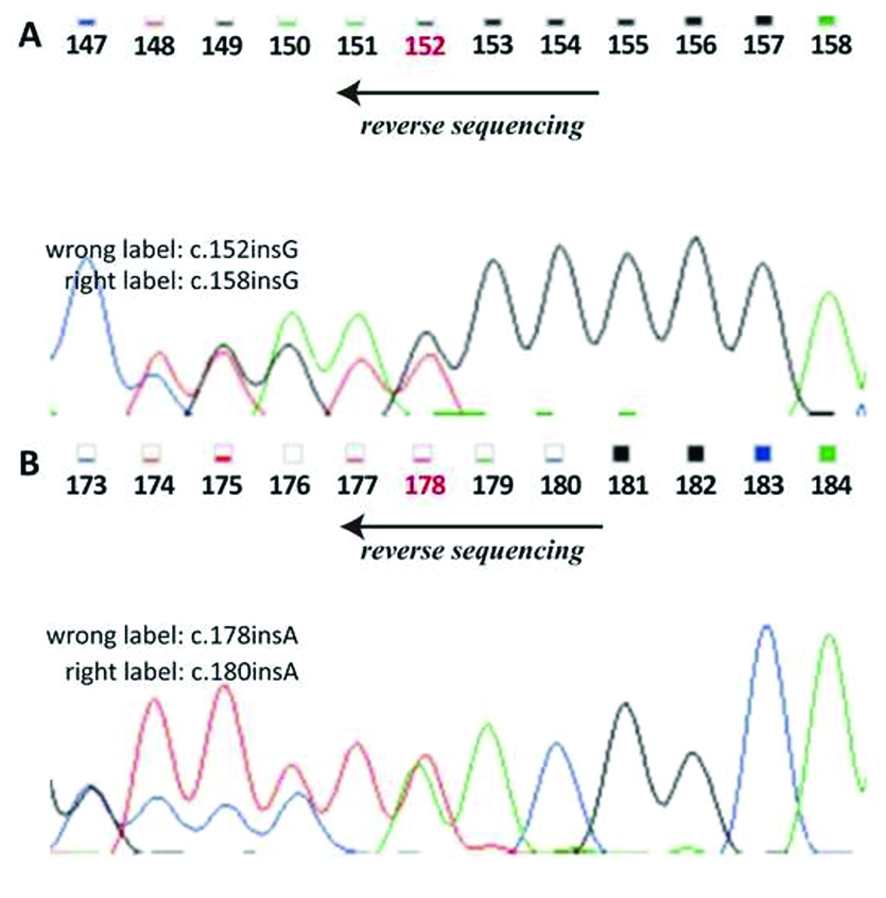
图2 c.158insG和c.180insA突变反向测序结果
Figure 2 Reverse sequencing results of c.158insG and c.180insA
3 讨 论
PJS是一种常染色体显性遗传病。分子遗传学研究表明,位于19号染色体短臂的STK11基因发生了杂合突变是该病的主要致病原因。Sanger测序联合MLPA或NGS联合MLPA是目前为止最好的组合检测策略[2]。研究显示,60%~78%PJS先证者的家属携带相同的致病性突变,而17%~40%先证者为突变病例[5]。一旦在先证者中发现STK11基因突变,其高危的亲属就需进行预测性基因突变检测。此外,STK11基因的突变检测有助于PJS的基因型-表型的研究,而该研究尤其对于恶性肿瘤风险的评估,具有重要的临床意义。STK11基因的编码区由9个外显子组成,其编码433个氨基酸的蛋白质,主要由三个结构域组成:N末端非催化结构域、催化激酶结构域和C末端非催化调节结构域。截止目前,在PJS或其他疾病患者中检测到的STK11突变可达412种,在全面回顾文献后,发现中国人群贡献了26.2%突变。在回顾文献同时,发现了一部分突变报道错误的情况,对此进行整理、归纳,大致可分为两大类:新发突变命名错误和已报道突变被重复报道。
新发突变命名错误的主要原因包括数据库收录不全和突变识别错误。中文期刊报道的三个突变,c.290+1G>T、c.109C>T和c.733C>T,因语言问题未被国外的数据库识别、收录[8,22]。在HGMD中记录的以上三个突变是被其他学者报道的,但该学者的研究出版时间晚于中国学者。而突变识别错误方面:Zheng等[16]报道了一例儿童患者的错义突变,命名为c.580G>T,并在文章中提到该突变先前被报道为PJS的新发致病突变。对文献交叉对比发现,研究报道的突变是c.580G>A,因此推断该情况是由于Zheng等将新发突变错误识别为已报道所致。而突变识别错误存在另外一种形式:Wang等[14]在2014年发现的c.891G>C突变,未被HGMD收录并宣布该突变为首次报道。赵晓等[11]早在2012年就对其进行了报道,但该文中未指出该突变为新发突变,且未被数据库收录。所以Wang等的误报是继发于赵晓等的识别错误。
导致突变报道错误的另一原因是将已报道突变误认为新发突变进行重复报道,原因包括:文献综述不足,因错误信息导致的后续错误报道,短期内重复报道以及因重复序列而命名错误。Zuo等[10]的c.180C>G,毛旭燕等[15]的c.180C>G、c.354C>G、c.521A>G、c.526G>A、c.862G>A,李蒙等[19]的c.298C>T、IVS4+1G>T、IVS8-2A>C、IVS1+1G>A、IVS7-2A>G,Zhang等[20]的c.598-1G>A,Wu等[21]的c.358G>T都被HGMD数据库收录,但是作者未进行全面、正确检索和识别,误认为是新发突变进而重复发表。短期内重复报道的主要原因是不同作者发现同一突变并短期内先后发表。如Wang等[14]在2014年报道了delexon3是一个新突变,但Ngeow等[24]在2013年已经报道过。相同情况同样发生在c.419T>C、IVS5-2A>G、c.243delG、c.716G>C这些突变上。短期内重复报道存在另外一种情况:同一作者在两篇不同文献[12]中均将c.469_498del30这一突变报道为新发突变。此外,当插入或缺失突变的碱基位于重复序列区域时,突变碱基的定位、命名容易发生错误。严格地说,该问题目前尚存争议,但常用方法是将重复序列最后一个核苷酸位点作为突变命名位点。相反,在重复序列的第一位核苷酸中的之前进行标记,并认为第一位核苷酸发生突变并不妥当。c.178insA,c.152insG实际上是命名错误,因为发生插入突变的位点应在第178~180位和第153~157位的重复序列之间。根据氨基酸的变化,正确的命名方式分别是c.179insA、c.180insA和c.153-158insG。为此对该PJS患者的STK11基因相应区域进行反向测序,并检测到这两个突变位点,并最终证实了正确命名方式为c.180insA和c.158insG。
在对PJS患者致病突变进行检测过程中,部分学者发现一个特殊现象:在同一位PJS患者的STK11基因上可检测到多个突变,便推断PJS患者可能同时存在多个致病突变。在缺少对突变致病性鉴定的情况下,该推论并不严谨。Tan等[25]在该方面的处理方式值得借鉴。该学者在两个不同的PJS家族中,检测到先证者同时携带两种突变c.890G>A、c.1062C>G和delexon1、c.1062C>G,先后通过突变数据库检索、家系和症状共分离情况、基因表达及功能试验、蛋白结构和功能预测软件的使用等多种方法证明,最终证实致病突变为c.890G>A和delexon1,而不是c.1062C>G。2015年美国医学遗传学学院和美国基因组学与分子病理学协会对于突变的报告方式,联合给出了详细的评定标准[26]。国内专家对其也进行了翻译,在国内进行广泛讨论和学习。我国学者可通过学习和实践该标准,对检出数据进行更加条理化分析,最终获得正确、有效的结果,并给予患者提供适当的家系检测和随访计划。
[1] Giardiello FM,Trimbath JD. Peutz-Jeghers syndrome and management recommendations[J]. Clin Gastroenterol Hepatol,2006,4(4):408-415.
[2] Wagner A,Aretz S,Auranen A,et al. The management of Peutz-Jeghers Syndrome:European hereditary tumour group(EHTG) guideline[J]. J Clin Med,2021,10(3):473.
[3] Tomlinson IP,Houlston RS. Peutz-Jeghers syndrome[J]. J Med Genet,1997,34(12):1007-1011.
[4] Beggs AD,Latchford AR,Vasen HF,et al. Peutz-Jeghers syndrome:a systematic review and recommendations for management[J]. Gut,2010,59(7):975-986.
[5] van Lier MG,Wagner A,Mathus-Vliegen EM,et al. High cancer risk in Peutz-Jeghers syndrome:a systematic review and surveillance recommendations[J]. Am J Gastroenterol,2010,105(6):1258-1264.
[6] Wu BD,Wang YJ,Fan LL,et al. Clinical and genetic analyses of 38 Chinese patients with Peutz-Jeghers syndrome[J]. Biomed Res Int,2020,2020:9159315.
[7] Daniell J,Plazzer JP,Perera A,et al. An exploration of genotype-phenotype link between Peutz-Jeghers syndrome and STK11:a review[J]. Fam Cancer,2018,17(3):421-427.
[8] 李宜雄,吕新生,夏家辉,等.中国人Peutz-Jeghers综合征STK11基因突变的研究[J].中华医学杂志,1999,79(6):425-427.
[9] 康连春,赵喜荣,周永双,等.STK11基因在Peutz-Jeghers综合征家系中的突变分析[J].科学通报,2002,47(21):1639-1643.
[10] Zuo YG,Xu KJ,Su B,et al. Two novel STK11 mutations in three Chinese families with Peutz-Jeghers syndrome[J]. Chin Med J(Engl),2007,120(13):1183-1186.
[11] 赵晓,李玉霞,凌焱,等.20例中国Peutz-Jeghers综合征患者STK11基因编码区突变分析[J].南方医科大学学报,2012,32(4):511-514.
[12] Chen C,Zhang X,Wang F,et al. One novel deletion and one splicing mutation of the LKB1 gene in two Chinese patients with Peutz-Jeghers Syndrome[J]. DNA Cell Biol,2012,31(10):1535-1540.
[13] Dai L,Fu L,Liu D,et al. Novel and recurrent mutations of STK11 gene in six Chinese cases with Peutz-Jeghers Syndrome[J]. Dig Dis Sci,2014,59(8):1856-1861.
[14] Wang Z,Wu B,Mosig RA,et al. STK11 domain XI mutations:candidate genetic drivers leading to the development of dysplastic polyps in Peutz-Jeghers Syndrome[J]. Hum Mutat,2014,35(7):851-858.
[15] 毛旭燕,张亚飞,毛高平,等.Peutz-Jeghers综合征患者STK11基因突变情况[J].世界华人消化杂志,2015,23(2):332-337.
[16] Zheng B,Wang C,Jia Z,et al. A clinical and molecular genetic study in 11 Chinese children with Peutz-Jeghers syndrome[J]. J Pediatr Gastroenterol Nutr,2017,64(4):559-564.
[17] 蒋宇亮,李伟聪,赵子夜,等.中国Peutz-Jeghers综合征患者STK11基因突变检测及高频突变分析[J].河北医科大学学报,2017,38(8):878-881,896.
[18] Zhao HM,Yang YJ,Duan JQ,et al. Clinical and genetic study of Children with Peutz-Jeghers Syndrome identifies a high frequency of STK11 de novo mutation[J]. J Pediatr Gastroenterol Nutr,2019,68(2):199-206.
[19] 李蒙,孙涛,蒋宇亮,等.64例Peutz-Jeghers综合征患者STK11基因变异分析[J].中华医学遗传学杂志,2019,36(9):862-865.
[20] Zhang Z,Duan FX,Gu GL,et al. Mutation analysis of related genes in hamartoma polyp tissue of Peutz-Jeghers syndrome[J]. World J Gastroenterol,2020,26(16):1926-1937.
[21] Wu BD,Wang YJ,Fan LL,et al. Clinical and genetic analyses of 38 Chinese patients with Peutz-Jeghers syndrome[J]. Bio Med Res Int,2020,2020:9159315.
[22] 王振军,严仲瑜,毕郭龙,等.国人黑斑息肉病LKB1基因胚系突变的检测[J].中华外科杂志,2000,38(2):104-105.
[23] Wang Z,Wu B,Mosig RA,et al. STK11 domain XI mutations:candidate genetic drivers leading to the development of dysplastic polyps in Peutz-Jeghers syndrome[J]. Hum Mutat,2014,35(7):851-858.
[24] Ngeow J,Heald B,Rybicki LA,et al. Prevalence of germline PTEN,BMPR1A,SMAD4,STK11,and ENG mutations in patients with moderate-load colorectal polyps[J]. Gastroenterology,2013,144(7):1402-1409.
[25] Tan H,Wei X,Yang P,et al. A lesson from a reported pathogenic variant in Peutz-Jeghers syndrome:a case report[J]. Fam Cancer,2017,16(3):417-422.
[26] Richards S,Aziz N,Bale S,et al. Standards and guidelines for the interpretation of sequence variants:a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology[J]. Genet Med,2015,17(5):405-424.